ຄວາມສະເພາະຂອງການກວດສອບ
ໃນກໍລະນີຫຼາຍທີ່ສຸດ, ຈຸດປະສົງຂອງການອອກແບບ primer ແມ່ນເພື່ອເພີ່ມປະສິດທິພາບສະເພາະຂອງ PCR.ນີ້ຖືກກໍານົດໂດຍອິດທິພົນທີ່ຄາດຄະເນຫຼາຍຫຼືຫນ້ອຍຂອງຕົວແປຫຼາຍ.ຕົວແປທີ່ສໍາຄັນອັນຫນຶ່ງແມ່ນລໍາດັບຢູ່ປາຍ 3′ຂອງ primer.
ສິ່ງສໍາຄັນ, ການວິເຄາະ PCR ທີ່ຖືກອອກແບບສໍາລັບສະເພາະແມ່ນມັກຈະຮັກສາປະສິດທິພາບສູງໃນໄລຍະການເຄື່ອນໄຫວທີ່ກວ້າງຂວາງ, ເພາະວ່າການວິເຄາະບໍ່ໄດ້ຜະລິດຜະລິດຕະພັນການຂະຫຍາຍທີ່ບໍ່ສະເພາະ, ດັ່ງນັ້ນຈຶ່ງສາມາດແຂ່ງຂັນກັບ PCR reagents ຫຼື inhibiting ຕິກິຣິຍາ amplification ຕົ້ນຕໍ.
ແນ່ນອນ, ໃນບາງກໍລະນີ, ຄວາມສະເພາະແມ່ນບໍ່ສໍາຄັນທີ່ສຸດ, ສໍາລັບຕົວຢ່າງ, ໃນເວລາທີ່ເປົ້າຫມາຍແມ່ນເພື່ອກໍານົດປະລິມານທີ່ກ່ຽວຂ້ອງຢ່າງໃກ້ຊິດແຕ່ເຊື້ອພະຍາດທີ່ແຕກຕ່າງກັນ, ການອອກແບບພິເສດ, ການເພີ່ມປະສິດທິພາບແລະມາດຕະຖານການກວດສອບແມ່ນຕ້ອງການ.
ເສັ້ນໂຄ້ງ melting ແມ່ນວິທີການມາດຕະຖານສໍາລັບການປະເມີນຄວາມສະເພາະຂອງ amplicons, ຢ່າງຫນ້ອຍໃນແງ່ຂອງການຂະຫຍາຍເປົ້າຫມາຍດຽວ.ຢ່າງໃດກໍ່ຕາມ, ມັນຕ້ອງໄດ້ຮັບການເນັ້ນຫນັກວ່າເສັ້ນໂຄ້ງ melting ສາມາດເຂົ້າໃຈຜິດເພາະວ່າ, ສໍາລັບການຍົກຕົວຢ່າງ, ພວກເຂົາສາມາດໄດ້ຮັບຜົນກະທົບຈາກຜົນກະທົບລວມຂອງ primers suboptimal ແລະຄວາມເຂັ້ມຂົ້ນຂອງແມ່ແບບຕ່ໍາ.
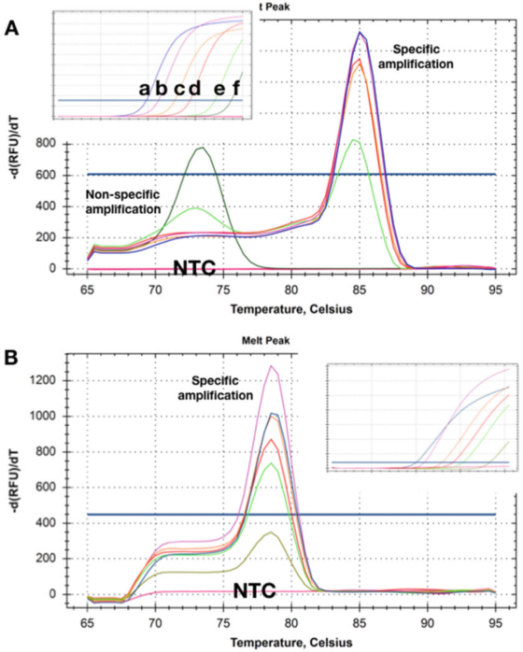
P5 |ເສັ້ນໂຄ້ງ melting ສະແດງໃຫ້ເຫັນການປ່ຽນແປງ Tm ທີ່ໄດ້ຮັບຈາກສອງການກວດຫາປະລິມານທີ່ແຕກຕ່າງກັນຂອງສອງ DNAs ເປົ້າຫມາຍ.
A. ຢູ່ທີ່ຄວາມເຂັ້ມຂຸ້ນທີ່ສູງຂຶ້ນ (ad)), ບໍ່ມີ primer dimer ຊັດເຈນຫຼັງຈາກການວັດແທກ qPCR ສໍາເລັດ.ເມື່ອຄວາມເຂັ້ມຂຸ້ນຂອງແມ່ແບບຫຼຸດລົງເຖິງ 50 ສໍາເນົາ (e), ຜະລິດຕະພັນທີ່ບໍ່ສະເພາະຈະປາກົດຂຶ້ນແລະກາຍເປັນຜະລິດຕະພັນດຽວທີ່ຄວາມເຂັ້ມຂົ້ນຕໍ່າສຸດ (f).
B. ການທົດສອບໄດ້ບັນທຶກ Tms ດຽວກັນຢູ່ໃນຄວາມເຂັ້ມຂຸ້ນຂອງເປົ້າຫມາຍທັງຫມົດ, ແລະບໍ່ມີ primer dimer ທີ່ຊັດເຈນເຖິງແມ່ນວ່າຢູ່ໃນຄວາມເຂັ້ມຂົ້ນຕ່ໍາສຸດ (5 ສໍາເນົາ).ເມື່ອນຳໃຊ້ວິທີການກວດຫາທັງສອງອັນນີ້, ບໍ່ມີຜະລິດຕະພັນການຂະຫຍາຍໃດຖືກກວດພົບໃນ NTCs.
P5 ສະແດງໃຫ້ເຫັນເສັ້ນໂຄ້ງການລະລາຍທີ່ໄດ້ຮັບກັບຕົວຢ່າງທີ່ແມ່ແບບແມ່ນມີຢູ່ໃນຄວາມເຂັ້ມຂຸ້ນທີ່ແຕກຕ່າງກັນ.P 5a ສະແດງໃຫ້ເຫັນວ່າຢູ່ໃນສອງຄວາມເຂັ້ມຂົ້ນຕ່ໍາສຸດ, Tms ຂອງຜະລິດຕະພັນການຂະຫຍາຍທີ່ບໍ່ສະເພາະທີ່ຜະລິດແມ່ນຕ່ໍາກວ່າຂອງ amplicons ສະເພາະ.
ແນ່ນອນ, ວິທີການກວດຫານີ້ບໍ່ສາມາດຖືກໃຊ້ຢ່າງເຊື່ອຖືໄດ້ເພື່ອກວດຫາເປົ້າໝາຍທີ່ມີຢູ່ໃນຄວາມເຂັ້ມຂຸ້ນຕໍ່າ.
ຫນ້າສົນໃຈ, NTCs, ie, ຕົວຢ່າງທີ່ບໍ່ມີ DNA ທັງຫມົດ, ບໍ່ໄດ້ບັນທຶກຜະລິດຕະພັນການຂະຫຍາຍ (ບໍ່ສະເພາະ), ເຊິ່ງຊີ້ໃຫ້ເຫັນວ່າ DNA genomic ພື້ນຖານສາມາດມີສ່ວນຮ່ວມໃນ amplification / polymerization ທີ່ບໍ່ສະເພາະ.
ບາງຄັ້ງ primers ພື້ນຖານດັ່ງກ່າວແລະການຂະຫຍາຍທີ່ບໍ່ສະເພາະແມ່ນບໍ່ສາມາດແກ້ໄຂໄດ້, ແຕ່ມັນມັກຈະເປັນໄປໄດ້ທີ່ຈະອອກແບບວິທີການກວດພົບທີ່ບໍ່ມີການຂະຫຍາຍທີ່ບໍ່ສະເພາະໃນຄວາມເຂັ້ມຂົ້ນຂອງແມ່ແບບໃດໆແລະ NTC (P 5b).
ໃນທີ່ນີ້, ເຖິງແມ່ນວ່າການບັນທຶກການຂະຫຍາຍຄວາມເຂັ້ມຂົ້ນຂອງເປົ້າຫມາຍທີ່ມີ Cq ຂອງ 35 ຈະຜະລິດເສັ້ນໂຄ້ງການລະລາຍສະເພາະ.ເຊັ່ນດຽວກັນ, NTCs ບໍ່ສະແດງອາການຂອງການຂະຫຍາຍທີ່ບໍ່ສະເພາະ.ບາງຄັ້ງ, ພຶດຕິກໍາການກວດພົບອາດຈະຂຶ້ນກັບເຫຼົ້າຂອງແມ່, ແລະພຽງແຕ່ກວດພົບການຂະຫຍາຍທີ່ບໍ່ສະເພາະຢູ່ໃນອົງປະກອບຂອງ buffer ບາງຢ່າງ, ເຊິ່ງອາດຈະກ່ຽວຂ້ອງກັບຄວາມເຂັ້ມຂົ້ນຂອງ Mg2+ ທີ່ແຕກຕ່າງກັນ.
ສະຖຽນລະພາບຂອງການກວດສອບ
ການເພີ່ມປະສິດທິພາບຂອງ Ta ແມ່ນຂັ້ນຕອນທີ່ເປັນປະໂຫຍດໃນຂະບວນການກວດສອບແລະເພີ່ມປະສິດທິພາບຂອງການກວດສອບ qPCR.ມັນສະຫນອງການຊີ້ບອກໂດຍກົງຂອງຄວາມຫມັ້ນຄົງຂອງ primer ທີ່ກໍານົດໄວ້ໂດຍການສະແດງອຸນຫະພູມ (ຫຼືລະດັບອຸນຫະພູມ) ທີ່ຜະລິດ Cq ຕ່ໍາສຸດໂດຍບໍ່ມີການຂະຫຍາຍ NTC.
ຄວາມແຕກຕ່າງລະຫວ່າງສອງຫາສີ່ເທົ່າໃນຄວາມອ່ອນໄຫວອາດຈະບໍ່ສໍາຄັນສໍາລັບຜູ້ທີ່ມີການສະແດງ mRNA ສູງ, ແຕ່ສໍາລັບການທົດສອບການວິນິດໄສ, ມັນອາດຈະຫມາຍເຖິງຄວາມແຕກຕ່າງລະຫວ່າງຜົນໄດ້ຮັບທາງລົບທາງບວກແລະທີ່ບໍ່ຖືກຕ້ອງ.
ຄຸນສົມບັດ Ta ຂອງ primers qPCR ອາດຈະແຕກຕ່າງກັນຢ່າງຫຼວງຫຼາຍ.ການທົດສອບບາງຢ່າງບໍ່ແຂງແຮງຫຼາຍ, ແລະຖ້າພວກເຂົາບໍ່ໄດ້ປະຕິບັດພາຍໃຕ້ມູນຄ່າ Ta ທີ່ດີທີ່ສຸດຂອງ primers, ພວກມັນຈະລົ້ມລົງຢ່າງໄວວາ.
ນີ້ແມ່ນສິ່ງສໍາຄັນເພາະວ່າການກວດພົບປະເພດນີ້ມັກຈະມີບັນຫາໃນໂລກທີ່ແທ້ຈິງ, ແລະຄວາມບໍລິສຸດຂອງຕົວຢ່າງ, ຄວາມເຂັ້ມຂົ້ນຂອງ DNA, ຫຼືການມີ DNA ອື່ນໆອາດຈະບໍ່ດີທີ່ສຸດ.
ນອກຈາກນັ້ນ, ຈໍານວນສໍາເນົາເປົ້າຫມາຍອາດຈະແຕກຕ່າງກັນໃນຂອບເຂດກ້ວາງ, ແລະ reagents, ເຄື່ອງໃຊ້ພາດສະຕິກ, ຫຼືເຄື່ອງມືອາດຈະແຕກຕ່າງຈາກການນໍາໃຊ້ໃນເວລາທີ່ຕັ້ງການທົດສອບ.
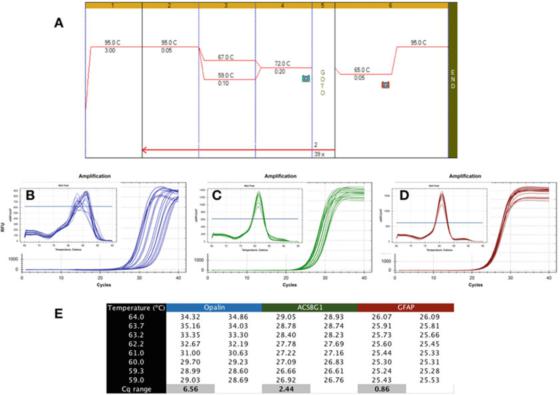
P6|ການປັບສີຂອງອຸນຫະພູມສະແດງໃຫ້ເຫັນຄວາມເຂັ້ມແຂງທີ່ແຕກຕ່າງກັນຂອງການກວດສອບ PCR.
A. ໃຊ້ Sensifast SYBR mastermix ຂອງ Bioline (ໝາຍເລກລາຍການ BIO-98050) ເພື່ອປະຕິບັດ PCR ກ່ຽວກັບ cDNA ທີ່ກະກຽມຈາກ RNA ສະຫມອງຂອງມະນຸດ.
B. ໃຊ້ເຄື່ອງມື CFX qPCR ຂອງ Bio-Rad ເພື່ອບັນທຶກແຜນທີ່ການຂະຫຍາຍ ແລະເສັ້ນໂຄ້ງການລະລາຍຂອງ apalene (NM_033207, F: GCCATGGAAGGAAAGTGACAGACC, R: CTCATGTGTGGGTGAATCTCCTAGG).
C. ເສັ້ນກຣາບການຂະຫຍາຍ ແລະເສັ້ນໂຄ້ງການລະລາຍຂອງ ACSBG1 (NM_015162.4, F: CTACACTTCCGGCACCACTGG, R: GTCCACGTGATATTGTCTTGACTCAG).
D. ກຣາບການຂະຫຍາຍ ແລະເສັ້ນໂຄ້ງການລະລາຍຂອງ GFAP (NM_002055.5, F: TGGAGAGGAAGATTGAGTCGCTGG, R: CGAACCTCCTCCTCGTGGATCTTC).
E. Cqs ບັນທຶກຢູ່ໃນອຸນຫະພູມ annealing ທີ່ແຕກຕ່າງກັນ, ສະແດງໃຫ້ເຫັນຄວາມແຕກຕ່າງຂອງ Cq ບັນທຶກພາຍໃຕ້ gradient ອຸນຫະພູມ 7C.
P 6 ສະແດງໃຫ້ເຫັນຜົນປົກກະຕິຂອງການທົດສອບທີ່ບໍ່ຕ້ອງການ, ບ່ອນທີ່ qPCR ໄດ້ຖືກປະຕິບັດໂດຍໃຊ້ gradient Tas ລະຫວ່າງ 59C ແລະ 67C (P 6a), ໂດຍໃຊ້ primers ສໍາລັບສາມພັນທຸກໍາຂອງສະຫມອງຂອງມະນຸດ.
ມັນສາມາດເຫັນໄດ້ຈາກເສັ້ນສະແດງການຂະຫຍາຍທີ່ opalin primers ແມ່ນຢູ່ໄກຈາກຄວາມເຫມາະສົມເພາະວ່າຊ່ວງ Ta ທີ່ດີທີ່ສຸດຂອງພວກເຂົາແມ່ນແຄບຫຼາຍ (ຮູບ 6b), ນັ້ນແມ່ນ, Cqs ໄດ້ຖືກກະແຈກກະຈາຍຢ່າງກວ້າງຂວາງ, ສົ່ງຜົນໃຫ້ Cqs ຖືກປຽບທຽບກັບ Cqs ຕ່ໍາທີ່ດີທີ່ສຸດຂອງພວກເຂົາ.
ວິທີການກວດພົບນີ້ບໍ່ຄົງທີ່ ແລະອາດຈະເຮັດໃຫ້ການຂະຫຍາຍໄດ້ດີທີ່ສຸດ.ດັ່ງນັ້ນ, ຄູ່ primers ນີ້ຄວນໄດ້ຮັບການອອກແບບໃຫມ່.ນອກຈາກນັ້ນ, ການວິເຄາະເສັ້ນໂຄ້ງ melting (inset) ສະແດງໃຫ້ເຫັນວ່າສະເພາະຂອງວິທີການກວດຫານີ້ອາດຈະມີບັນຫາ, ເນື່ອງຈາກວ່າເສັ້ນໂຄ້ງ melting ຂອງແຕ່ລະ Ta ແມ່ນແຕກຕ່າງກັນ.
ວິທີການກວດຫາ ACSBG1 ທີ່ສະແດງຢູ່ໃນ P 6c ແມ່ນເຂັ້ມແຂງກວ່າວິທີການກວດຫາ Opalin ຂ້າງເທິງ, ແຕ່ມັນຍັງຢູ່ໄກຈາກທີ່ເຫມາະສົມ, ແລະມັນເປັນໄປໄດ້ວ່າມັນສາມາດປັບປຸງໄດ້.
ຢ່າງໃດກໍ່ຕາມ, ພວກເຮົາເນັ້ນຫນັກວ່າບໍ່ມີການເຊື່ອມຕໍ່ທີ່ຈໍາເປັນລະຫວ່າງຄວາມທົນທານແລະຄວາມສະເພາະ, ເພາະວ່າເສັ້ນໂຄ້ງການລະລາຍທີ່ຜະລິດໂດຍວິທີການກວດພົບນີ້ສະແດງໃຫ້ເຫັນເຖິງມູນຄ່າສູງສຸດດຽວກັນໃນ Tas (inset).
ໃນທາງກົງກັນຂ້າມ, ການທົດສອບຄວາມທົນທານແມ່ນມີຄວາມທົນທານຫຼາຍ, ການຜະລິດ Cqs ທີ່ຄ້າຍຄືກັນໃນລະດັບຄວາມກ້ວາງຂອງ Tas, ໃນການທົດສອບ GFAP ສະແດງໃຫ້ເຫັນໃນ P 6d.
ຄວາມແຕກຕ່າງຂອງ Cqs ທີ່ໄດ້ຮັບໃນລະດັບດຽວກັນ 8 ອົງສາເຊນຊຽດແມ່ນຫນ້ອຍກວ່າ 1, ແລະເສັ້ນໂຄ້ງການລະລາຍ (inset) ຢືນຢັນລັກສະນະການກວດພົບໃນລະດັບອຸນຫະພູມນີ້.ມັນເປັນມູນຄ່າທີ່ສັງເກດວ່າ Tas ທີ່ຄິດໄລ່ແລະລະດັບ Ta ຕົວຈິງອາດຈະແຕກຕ່າງກັນຫຼາຍ.
ມີຄໍາແນະນໍາຫຼາຍຢ່າງທີ່ຖືກອອກແບບມາເພື່ອຊ່ວຍໃຫ້ນັກຄົ້ນຄວ້າອອກແບບ primers ທີ່ມີປະສິດທິພາບ, ສ່ວນຫຼາຍແມ່ນອີງໃສ່ກົດລະບຽບທີ່ມີມາດົນນານແລະຄວາມສົນໃຈຫຼາຍແມ່ນໄດ້ຖືກຈ່າຍໃຫ້ກັບ 3′ຂອງ primers.ມັນມັກຈະແນະນໍາໃຫ້ປະກອບມີ G ຫຼື C ຢູ່ປາຍ 3' ແລະສອງຖານ G ຫຼື C (GC clamp), ແຕ່ບໍ່ເກີນສອງຖານຂອງ 5 ສຸດທ້າຍ.
ໃນການປະຕິບັດ, ກົດລະບຽບເຫຼົ່ານີ້ສາມາດນໍາພານັກຄົ້ນຄວ້າ, ແຕ່ພວກເຂົາບໍ່ຈໍາເປັນຕ້ອງຖືກຕ້ອງໃນທຸກສະຖານະການ.
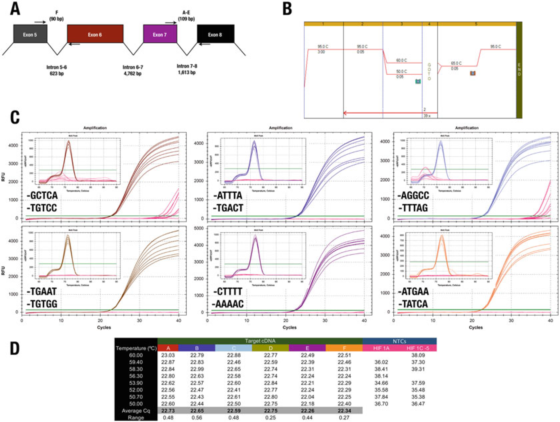
P7 |3′ປາຍຂອງ primer ມີຜົນກະທົບພຽງເລັກນ້ອຍກ່ຽວກັບສະເພາະຫຼືປະສິດທິພາບ.
A. ຕໍາແຫນ່ງຂອງ primers ສໍາລັບ gene HIF-1α (NM_181054.2) ຂອງມະນຸດ.
B. ໃຊ້ Agilent Brilliant III SYBR ເຫຼົ້າແມ່ສີຂຽວ (Cat. No. 600882) ເພື່ອຂະຫຍາຍ 6 ລາຍການທົດສອບ.
C. ການຂະຫຍາຍເສັ້ນສະແດງ ແລະເສັ້ນໂຄ້ງການລະລາຍທີ່ບັນທຶກໂດຍເຄື່ອງມື CFX qPCR ຂອງ Bio-Rad ແລະ 3′ primers.NTCs ສະແດງໃຫ້ເຫັນເປັນສີແດງ.
D. ບັນທຶກ Cqs ຂອງແຕ່ລະລາຍການທົດສອບ
ຕົວຢ່າງ, ຜົນໄດ້ຮັບໃນ P 7 ກົງກັນຂ້າມກັບກົດລະບຽບ 3′.ການອອກແບບທັງຫມົດຜະລິດຜົນໄດ້ຮັບດຽວກັນໂດຍພື້ນຖານ, ມີພຽງແຕ່ສອງປະສົມປະສານ primer ນໍາໄປສູ່ການຂະຫຍາຍທີ່ບໍ່ສະເພາະໃນ NTC.
ຢ່າງໃດກໍຕາມ, ພວກເຮົາບໍ່ສາມາດສະຫນັບສະຫນູນຜົນກະທົບຂອງ clip GC, ເນື່ອງຈາກວ່າໃນກໍລະນີນີ້, ການນໍາໃຊ້ A ຫຼື T ເປັນສູງສຸດຂອງ 30 ພື້ນຖານບໍ່ໄດ້ຫຼຸດລົງສະເພາະ.
ການທົດສອບ C, ບ່ອນທີ່ F primer ສິ້ນສຸດລົງໃນ GGCC, ໄດ້ບັນທຶກ Cqs ໃນ NTCs, ຊີ້ໃຫ້ເຫັນວ່າຫນຶ່ງອາດຈະຕ້ອງການທີ່ຈະຫລີກລ້ຽງລໍາດັບເຫຼົ່ານີ້ຢູ່ໃນ 30-end.ພວກເຮົາເນັ້ນຫນັກວ່າວິທີດຽວທີ່ຈະກໍານົດລໍາດັບ 3'end ທີ່ດີທີ່ສຸດຂອງຄູ່ primer ແມ່ນການປະເມີນຜູ້ສະຫມັກ primers ບາງຄົນໃນການທົດລອງ.
ປະສິດທິພາບການຂະຫຍາຍ
ສິ່ງສໍາຄັນ, ເຖິງແມ່ນວ່າການກວດຫາ PCR ທີ່ບໍ່ສະເພາະບໍ່ສາມາດກາຍເປັນສະເພາະ, ປະສິດທິພາບການຂະຫຍາຍສາມາດຖືກປັບແລະສູງສຸດໃນຫຼາຍວິທີທີ່ແຕກຕ່າງກັນໂດຍການປ່ຽນ enzyme, ເຫຼົ້າແມ່, ທາດເສີມ, ແລະເງື່ອນໄຂການຂີ່ລົດຖີບ.
ເພື່ອປະເມີນປະສິດທິພາບຂອງການກວດຫາ PCR, ມັນດີທີ່ສຸດທີ່ຈະໃຊ້ການເຈືອຈາງ serial ຂອງ 10 ຫຼື 5 ເທົ່າຂອງອາຊິດ nucleic ເປົ້າຫມາຍ, ນັ້ນແມ່ນ, "ວິທີການເສັ້ນໂຄ້ງມາດຕະຖານ".
ຖ້າ PCR amplicons ຫຼືເປົ້າຫມາຍ DNA ສັງເຄາະຖືກນໍາໃຊ້ເພື່ອສ້າງເສັ້ນໂຄ້ງມາດຕະຖານ, ການເຈືອຈາງ serial ຂອງເປົ້າຫມາຍເຫຼົ່ານີ້ຄວນໄດ້ຮັບການປະສົມກັບຈໍານວນຄົງທີ່ຂອງ DNA ພື້ນຖານ (ເຊັ່ນ DNA ພັນທຸ ກຳ).
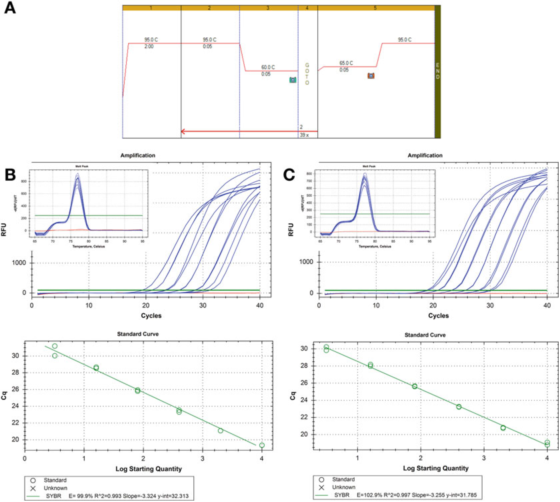
P8 |Dilution curve ເພື່ອປະເມີນປະສິດທິພາບຂອງ PCR.
A. ໃຊ້ primers ສໍາລັບ HIF-1: F: AAGAACTTTTAGGCCGCTCA ແລະ R: TGTCCTGTGGTGACTTGTCC ແລະ Agilent's Brilliant III SYBR Green mastermix (ຈໍານວນລາຍການ 600882) ສໍາລັບ PCR ແລະສະພາບເສັ້ນໂຄ້ງ melting.
B. 100 ng RNA ຖືກຖອດຂໍ້ຄວາມແບບປີ້ນກັບກັນ, ເຈືອຈາງ 2 ເທື່ອ, ແລະຕົວຢ່າງ cDNA ທີ່ເຈືອຈາງຕາມລໍາດັບໄດ້ຖືກເຈືອຈາງ 5 ເທື່ອເປັນ 1 ng DNA ພັນທຸ ກຳ ຂອງມະນຸດ.ເສັ້ນໂຄ້ງ melting ແມ່ນສະແດງໃຫ້ເຫັນໃນ inset ໄດ້.
C. ປະຕິກິລິຍາ RT, ການເຈືອຈາງແລະການເຈືອຈາງ serial ແມ່ນຊ້ໍາກັນສໍາລັບຕົວຢ່າງ cDNA ທີສອງ, ແລະຜົນໄດ້ຮັບແມ່ນຄ້າຍຄືກັນ.
P 8 ສະແດງໃຫ້ເຫັນເສັ້ນໂຄ້ງມາດຕະຖານສອງຢ່າງ, ໂດຍນໍາໃຊ້ວິທີການກວດພົບດຽວກັນໃນສອງຕົວຢ່າງ cDNA ທີ່ແຕກຕ່າງກັນ, ຜົນໄດ້ຮັບແມ່ນປະສິດທິພາບດຽວກັນ, ປະມານ 100%, ແລະຄ່າ R2 ຍັງຄ້າຍຄືກັນ, ນັ້ນແມ່ນ, ລະດັບຄວາມສອດຄ່ອງລະຫວ່າງຂໍ້ມູນການທົດລອງແລະເສັ້ນ regression ຫຼືຂໍ້ມູນລະດັບຂອງ linearity.
ສອງເສັ້ນໂຄ້ງມາດຕະຖານແມ່ນປຽບທຽບໄດ້, ແຕ່ບໍ່ຄືກັນ.ຖ້າຈຸດປະສົງແມ່ນເພື່ອກໍານົດປະລິມານເປົ້າຫມາຍຢ່າງຖືກຕ້ອງ, ມັນຕ້ອງສັງເກດວ່າມັນບໍ່ສາມາດຍອມຮັບໄດ້ທີ່ຈະສະຫນອງການຄິດໄລ່ຕົວເລກສໍາເນົາໂດຍບໍ່ມີການອະທິບາຍຄວາມບໍ່ແນ່ນອນ.
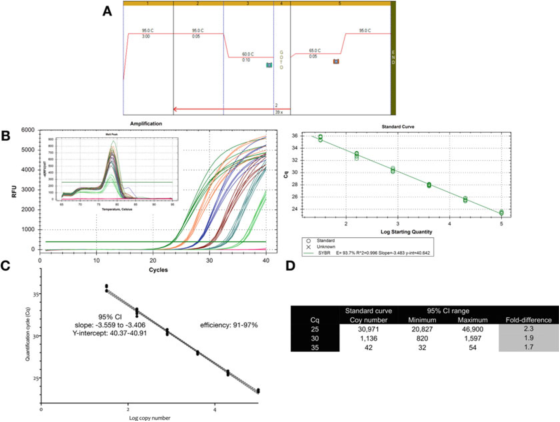
P9 |ຄວາມບໍ່ແນ່ນອນຂອງການວັດແທກທີ່ກ່ຽວຂ້ອງກັບປະລິມານໂດຍໃຊ້ເສັ້ນໂຄ້ງມາດຕະຖານ.
A. ໃຊ້ primers ສໍາລັບ GAPDH (NM_002046) ເພື່ອປະຕິບັດສະພາບເສັ້ນໂຄ້ງຂອງ PCR ແລະ melting.F: ACAGTTGCCATGTAGACC ແລະ R: TAACTGGTTGAGCAGG ແລະ Sensifast SYBR mastermix ຂອງ Bioline (ໝາຍເລກລາຍການ BIO-98050).
B. ຕາຕະລາງການຂະຫຍາຍ, ເສັ້ນໂຄ້ງ melting ແລະເສັ້ນໂຄ້ງມາດຕະຖານທີ່ບັນທຶກໄວ້ດ້ວຍເຄື່ອງມື CFX qPCR ຂອງ Bio-Rad.
C. ເສັ້ນໂຄ້ງມາດຕະຖານ ແລະ ໄລຍະຄວາມເຊື່ອໝັ້ນ 95% (CI).
D. ຈໍານວນສໍາເນົາແລະ 95% ໄລຍະເວລາຄວາມຫມັ້ນໃຈຂອງສາມຄ່າ Cq ໄດ້ມາຈາກເສັ້ນໂຄ້ງ dilution.
P 9 ສະແດງໃຫ້ເຫັນວ່າສໍາລັບການທົດສອບທີ່ດີທີ່ສຸດ, ການປ່ຽນແປງທີ່ປະກົດຕົວຂອງເສັ້ນໂຄ້ງມາດຕະຖານດຽວແມ່ນປະມານ 2 ເທົ່າ (ໄລຍະຄວາມຫມັ້ນໃຈ 95%, ຕ່ໍາສຸດເຖິງສູງສຸດ), ເຊິ່ງອາດຈະເປັນຕົວແປທີ່ນ້ອຍທີ່ສຸດທີ່ສາມາດຄາດຫວັງໄດ້.
ຜະລິດຕະພັນທີ່ກ່ຽວຂ້ອງ:
ເວລາປະກາດ: ກັນຍາ-30-2021