1. ຄວາມຮູ້ພື້ນຖານ (ຖ້າທ່ານຕ້ອງການເບິ່ງພາກທົດລອງ, ກະລຸນາໂອນໂດຍກົງກັບພາກທີສອງ)
ໃນຖານະເປັນປະຕິກິລິຍາ derivative ຂອງ PCR ທໍາມະດາ, ເວລາຈິງ PCR ສ່ວນໃຫຍ່ແມ່ນຕິດຕາມກວດກາການປ່ຽນແປງຂອງປະລິມານຂອງຜະລິດຕະພັນ amplification ໃນແຕ່ລະວົງຈອນຂອງປະຕິກິລິຍາການຂະຫຍາຍ PCR ໃນເວລາທີ່ແທ້ຈິງໂດຍຜ່ານການປ່ຽນແປງຂອງສັນຍານ fluorescence, ແລະການວິເຄາະປະລິມານຂອງແມ່ແບບເລີ່ມຕົ້ນໂດຍຜ່ານຄວາມສໍາພັນລະຫວ່າງຄ່າ ct ແລະເສັ້ນໂຄ້ງມາດຕະຖານ.
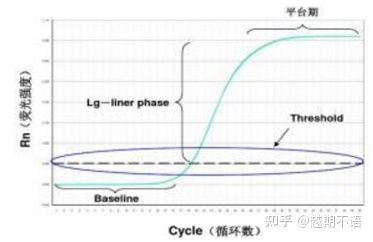
ຂໍ້ມູນສະເພາະຂອງ RT-PCR ແມ່ນພື້ນຖານ, ເກນ fluorescenceແລະຄ່າ Ct.
| ພື້ນຖານ: | ຄ່າ fluorescence ຂອງ 3rd-15th cycle ແມ່ນພື້ນຖານ (baseline), ເຊິ່ງເກີດມາຈາກຄວາມຜິດພາດຂອງການວັດແທກເປັນບາງຄັ້ງຄາວ. |
| ເກນ (threshold): | ຫມາຍເຖິງການກໍານົດຂອບເຂດການກວດພົບ fluorescence ທີ່ຕັ້ງຢູ່ໃນຕໍາແຫນ່ງທີ່ເຫມາະສົມໃນພາກພື້ນການຂະຫຍາຍຕົວຂອງເສັ້ນໂຄ້ງຂະຫຍາຍໃຫຍ່ຂື້ນ, ໂດຍທົ່ວໄປແລ້ວ 10 ເທົ່າຂອງຄ່າບ່ຽງເບນມາດຕະຖານຂອງເສັ້ນພື້ນຖານ. |
| ຄ່າ CT: | ມັນແມ່ນຈໍານວນຂອງວົງຈອນ PCR ເມື່ອຄ່າ fluorescence ໃນແຕ່ລະທໍ່ປະຕິກິລິຍາເຖິງເກນ. ຄ່າ Ct ແມ່ນອັດຕາສ່ວນກົງກັນຂ້າມກັບປະລິມານຂອງແມ່ແບບເບື້ອງຕົ້ນ. |
ວິທີການຕິດສະຫຼາກທົ່ວໄປສໍາລັບ RT-PCR:
| ວິທີການ | ປະໂຫຍດ | ຂໍ້ບົກຜ່ອງ | ຂອບເຂດຂອງຄໍາຮ້ອງສະຫມັກ |
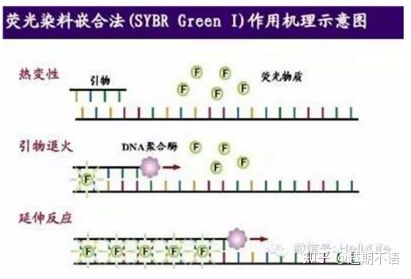
| SYBR ສີຂຽວⅠ | ການນໍາໃຊ້ທີ່ກວ້າງຂວາງ, ລະອຽດອ່ອນ, ລາຄາຖືກແລະສະດວກສະບາຍ | ຄວາມຕ້ອງການ primer ແມ່ນສູງ, ມັກຈະເປັນແຖບທີ່ບໍ່ແມ່ນສະເພາະ | ມັນເຫມາະສົມສໍາລັບການວິເຄາະປະລິມານຂອງ genes ເປົ້າຫມາຍຕ່າງໆ, ການຄົ້ນຄວ້າກ່ຽວກັບການສະແດງອອກຂອງ gene, ແລະການຄົ້ນຄວ້າກ່ຽວກັບສັດແລະພືດ recombinant transgenic. |
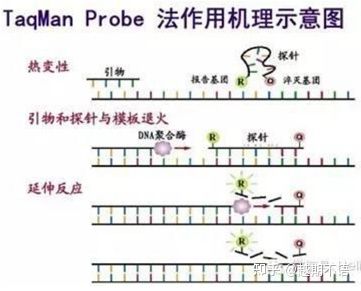
| ຕາກແມນ | ສະເພາະທີ່ດີ ແລະສາມາດເຮັດຊ້ຳໄດ້ສູງ | ລາຄາແມ່ນສູງແລະພຽງແຕ່ເຫມາະສົມສໍາລັບເປົ້າຫມາຍສະເພາະ. | ການກວດຫາເຊື້ອພະຍາດ, ການຄົ້ນຄວ້າ gene ການຕໍ່ຕ້ານຢາເສບຕິດ, ການປະເມີນປະສິດທິພາບຂອງຢາ, ການວິນິດໄສພະຍາດທາງພັນທຸກໍາ. |
| beacon ໂມເລກຸນ | ຄວາມສະເພາະສູງ, fluorescence, ພື້ນຫລັງຕ່ໍາ | ລາຄາແມ່ນສູງ, ມັນເຫມາະສົມສໍາລັບຈຸດປະສົງສະເພາະໃດຫນຶ່ງ, ການອອກແບບແມ່ນມີຄວາມຫຍຸ້ງຍາກ, ແລະລາຄາແມ່ນສູງ. | ການວິເຄາະ gene ສະເພາະ, ການວິເຄາະ SNP |
2. ຂັ້ນຕອນການທົດລອງ
2.1 ກ່ຽວກັບການຈັດກຸ່ມທົດລອງ- ຕ້ອງມີນໍ້າສ້າງຫຼາຍຂຸມໃນກຸ່ມ, ແລະຕ້ອງມີການຊໍ້າຄືນທາງຊີວະພາບ.
| ① | ການຄວບຄຸມເປົ່າ | ໃຊ້ເພື່ອກວດຫາສະຖານະການຂະຫຍາຍຕົວຂອງເຊນໃນການທົດລອງ |
| ② | ການຄວບຄຸມທາງລົບ siRNA (ລໍາດັບ siRNA ທີ່ບໍ່ສະເພາະ) | ສະແດງໃຫ້ເຫັນສະເພາະຂອງການປະຕິບັດ RNAi.siRNA ອາດຈະເຮັດໃຫ້ເກີດການຕອບສະຫນອງຄວາມກົດດັນທີ່ບໍ່ສະເພາະທີ່ຄວາມເຂັ້ມຂຸ້ນຂອງ 200nM. |
| ③ | Transfection Reagent ການຄວບຄຸມ | ບໍ່ລວມເຖິງຄວາມເປັນພິດຂອງທາດປະຕິການຖ່າຍທອດຕໍ່ຈຸລັງ ຫຼືຜົນກະທົບຕໍ່ການສະແດງອອກຂອງ gene ເປົ້າໝາຍ. |
| ④ | siRNA ຕໍ່ gene ເປົ້າຫມາຍ | ລົບການສະແດງອອກຂອງ gene ເປົ້າຫມາຍ |
| ⑤ (ທາງເລືອກ) | siRNA ບວກ | ໃຊ້ເພື່ອແກ້ໄຂບັນຫາລະບົບການທົດລອງແລະບັນຫາການດໍາເນີນງານ |
| ⑥ (ທາງເລືອກ) | ການຄວບຄຸມ fluorescent siRNA | ປະສິດທິພາບຂອງການຖ່າຍທອດຈຸລັງສາມາດສັງເກດໄດ້ດ້ວຍກ້ອງຈຸລະທັດ |
2.2 ຫຼັກການຂອງການອອກແບບ primer
| ຂະຫຍາຍຂະໜາດຊິ້ນສ່ວນ | ມັກຢູ່ທີ່ 100-150bp |
| ຄວາມຍາວຂອງ primer | 18-25bp |
| ເນື້ອໃນ GC | 30%-70%, ມັກ 45%-55% |
| ຄ່າ Tm | 58-60℃ |
| ລໍາດັບ | ຫຼີກເວັ້ນການ T / C ຢ່າງຕໍ່ເນື່ອງ;A/G ຢ່າງຕໍ່ເນື່ອງ |
| 3 ລໍາດັບທ້າຍ | ຫຼີກເວັ້ນການ GC ອຸດົມສົມບູນຫຼື AT ອຸດົມສົມບູນ;ພື້ນຖານຂອງ terminal ແມ່ນມັກ G ຫຼື C;ມັນດີທີ່ສຸດທີ່ຈະຫຼີກເວັ້ນ T |
| ຄວາມສົມບູນ | ຫຼີກລ້ຽງການຈັດລໍາດັບທີ່ສົມທຽບກັນຫຼາຍກວ່າ 3 ຖານພາຍໃນ primer ຫຼືລະຫວ່າງສອງ primers |
| ສະເພາະ | ໃຊ້ການຄົ້ນຫາລະເບີດເພື່ອຢືນຢັນຄວາມສະເພາະຂອງ primer |
①SiRNA ແມ່ນສະເພາະຊະນິດ, ແລະລໍາດັບຂອງຊະນິດພັນຕ່າງໆຈະແຕກຕ່າງກັນ.
②SiRNA ຖືກຫຸ້ມຫໍ່ຢູ່ໃນຝຸ່ນແຫ້ງ freeze, ເຊິ່ງສາມາດເກັບຮັກສາໄວ້ຢ່າງຫມັ້ນຄົງສໍາລັບ 2-4 ອາທິດໃນອຸນຫະພູມຫ້ອງ.
2.3 ເຄື່ອງມືຫຼືທາດປະສົມທີ່ຕ້ອງໄດ້ຮັບການກະກຽມລ່ວງຫນ້າ
| Primer (ເອກະສານອ້າງອີງພາຍໃນ) | ລວມທັງໄປຂ້າງຫນ້າແລະປີ້ນກັບສອງ |
| Primers (ພັນທຸກໍາເປົ້າຫມາຍ) | ລວມທັງໄປຂ້າງຫນ້າແລະປີ້ນກັບສອງ |
| ເປົ້າໝາຍ Si RNA (3 ແຖບ) | ໂດຍທົ່ວໄປແລ້ວ, ບໍລິສັດຈະສັງເຄາະ 3 ແຖບ, ແລະຫຼັງຈາກນັ້ນເລືອກເອົາຫນຶ່ງໃນສາມໂດຍ RT-PCR. |
| ຊຸດຖ່າຍໂອນ | Lipo2000 ແລະອື່ນໆ. |
| ຊຸດສະກັດ RNA ຢ່າງໄວວາ | ສໍາລັບການສະກັດ RNA ຫຼັງຈາກ transfection |
| ຊຸດການຖອດຂໍ້ຄວາມແບບປີ້ນກັບກັນຢ່າງໄວວາ | ສໍາລັບການສັງເຄາະ cDNA |
| ຊຸດຂະຫຍາຍ PCR | 2× Super SYBR ສີຂຽວ qPCR Master Mix |
2.4 ກ່ຽວກັບບັນຫາທີ່ຕ້ອງໄດ້ເອົາໃຈໃສ່ໃນຂັ້ນຕອນການທົດລອງສະເພາະ:
①siRNA ຂະບວນການຖ່າຍທອດ
1. ສໍາລັບແຜ່ນ, ທ່ານສາມາດເລືອກແຜ່ນ 24 ດີ, ແຜ່ນ 12 ດີຫຼື 6 ແຜ່ນ (ຄວາມເຂັ້ມຂົ້ນຂອງ RNA ສະເລ່ຍທີ່ສະເຫນີໃນແຕ່ລະຂຸມຂອງ 24 ດີແມ່ນປະມານ 100-300 ng / uL), ແລະຄວາມຫນາແຫນ້ນຂອງການຖ່າຍທອດທີ່ດີທີ່ສຸດຂອງຈຸລັງແມ່ນສູງເຖິງ 60% -80% ຫຼືຫຼາຍກວ່ານັ້ນ.
2. ຂັ້ນຕອນການຖ່າຍທອດ ແລະ ຄວາມຕ້ອງການສະເພາະແມ່ນປະຕິບັດຕາມຄໍາແນະນໍາຢ່າງເຂັ້ມງວດ.
3. ຫຼັງຈາກຖ່າຍທອດ, ຕົວຢ່າງສາມາດເກັບໄດ້ພາຍໃນ 24-72 ຊົ່ວໂມງເພື່ອກວດຫາ mRNA (RT-PCR) ຫຼືການກວດທາດໂປຼຕີນພາຍໃນ 48-96 ຊົ່ວໂມງ (WB)
② ຂະບວນການສະກັດ RNA
1. ປ້ອງກັນການປົນເປື້ອນໂດຍ enzymes exogenous.ມັນສ່ວນໃຫຍ່ແມ່ນໃສ່ຫນ້າກາກແລະຖົງມືຢ່າງເຂັ້ມງວດ;ການນໍາໃຊ້ຄໍາແນະນໍາ pipette sterilized ແລະທໍ່ EP;ນ້ໍາທີ່ໃຊ້ໃນການທົດລອງຕ້ອງເປັນ RNase-Free.
2. ແນະນໍາໃຫ້ເຮັດສອງຄັ້ງຕາມຄໍາແນະນໍາໃນຊຸດສະກັດໄວ, ເຊິ່ງຈະຊ່ວຍປັບປຸງຄວາມບໍລິສຸດແລະຜົນຜະລິດຢ່າງແທ້ຈິງ.
3. ນໍ້າເສຍຈະຕ້ອງບໍ່ແຕະຖັນ RNA.
③ ປະລິມານ RNA
ຫຼັງຈາກ RNA ໄດ້ຖືກສະກັດ, ມັນສາມາດຖືກຄິດໄລ່ໂດຍກົງດ້ວຍ Nanodrop, ແລະການອ່ານຂັ້ນຕ່ໍາສາມາດຕ່ໍາເຖິງ 10ng / ul.
④ ຂະບວນການຖອດຖອນຄືນ
1. ເນື່ອງຈາກຄວາມອ່ອນໄຫວສູງຂອງ RT-qPCR, ຢ່າງຫນ້ອຍ 3 ຂຸມຂະຫນານຄວນເຮັດສໍາລັບແຕ່ລະຕົວຢ່າງເພື່ອປ້ອງກັນບໍ່ໃຫ້ Ct ຕໍ່ມາບໍ່ແຕກຕ່າງກັນຫຼື SD ຂະຫນາດໃຫຍ່ເກີນໄປສໍາລັບການວິເຄາະສະຖິຕິ.
2. ຫ້າມແຊ່ແຂງ ແລະ ທາປະສົມ Master ຊ້ຳໆ.
3. ແຕ່ລະທໍ່ / ຂຸມຕ້ອງໄດ້ຮັບການທົດແທນດ້ວຍປາຍໃຫມ່!ຢ່າໃຊ້ທໍ່ທໍ່ດຽວກັນຢ່າງຕໍ່ເນື່ອງເພື່ອເພີ່ມຕົວຢ່າງ!
4. ຮູບເງົາທີ່ຕິດຢູ່ກັບແຜ່ນ 96 ດີຫຼັງຈາກເພີ່ມຕົວຢ່າງຕ້ອງໄດ້ຮັບການຂັດດ້ວຍແຜ່ນ.ມັນເປັນສິ່ງທີ່ດີທີ່ສຸດທີ່ຈະ centrifuge ກ່ອນທີ່ຈະເອົາໃສ່ເຄື່ອງ, ເພື່ອໃຫ້ຂອງແຫຼວໃນຝາທໍ່ສາມາດໄຫຼລົງແລະເອົາຟອງອາກາດ.
⑤ການວິເຄາະເສັ້ນໂຄ້ງທົ່ວໄປ
| ບໍ່ມີໄລຍະເວລາຂອງການຂະຫຍາຍຕົວຂອງ logarithmic | ອາດຈະມີຄວາມເຂັ້ມຂຸ້ນສູງຂອງແມ່ແບບ |
| ບໍ່ມີຄ່າ CT | ຂັ້ນຕອນທີ່ບໍ່ຖືກຕ້ອງສໍາລັບການກວດສອບສັນຍານ fluorescent; ການເຊື່ອມໂຊມຂອງ primers ຫຼື probes - ຄວາມສົມບູນຂອງມັນສາມາດກວດພົບໄດ້ໂດຍ PAGE electrophoresis; ຈໍານວນບໍ່ພຽງພໍຂອງແມ່ແບບ; ການເສື່ອມສະພາບຂອງແມ່ແບບ - ຫຼີກເວັ້ນການນໍາເອົາສິ່ງເສດເຫຼືອແລະການແຊ່ແຂໍງຊ້ໍາແລະ thawing ໃນການກະກຽມຕົວຢ່າງ; |
| Ct>38 | ປະສິດທິພາບການຂະຫຍາຍຕ່ໍາ;ຜະລິດຕະພັນ PCR ຍາວເກີນໄປ;ອົງປະກອບປະຕິກິລິຍາຕ່າງໆຖືກຊຸດໂຊມ |
| ເສັ້ນໂຄ້ງການຂະຫຍາຍເສັ້ນ | Probes ອາດຈະຖືກທໍາລາຍບາງສ່ວນໂດຍຮອບວຽນຂອງ freeze-thaw ຊ້ໍາຊ້ອນຫຼືການສໍາຜັດກັບແສງດົນນານ. |
| ຄວາມແຕກຕ່າງໃນຂຸມທີ່ຊ້ໍາກັນແມ່ນຂະຫນາດໃຫຍ່ໂດຍສະເພາະ | ການແກ້ໄຂປະຕິກິລິຍາບໍ່ໄດ້ຖືກລະລາຍຢ່າງສົມບູນຫຼືການແກ້ໄຂປະຕິກິລິຢາບໍ່ໄດ້ຖືກປະສົມ;ອາບນ້ໍາຄວາມຮ້ອນຂອງເຄື່ອງມື PCR ຖືກປົນເປື້ອນໂດຍສານ fluorescent |
2.5 ກ່ຽວກັບການວິເຄາະຂໍ້ມູນ
ການວິເຄາະຂໍ້ມູນຂອງ qPCR ສາມາດແບ່ງອອກເປັນປະລິມານທີ່ກ່ຽວຂ້ອງແລະປະລິມານຢ່າງແທ້ຈິງ.ຕົວຢ່າງ, ຈຸລັງໃນກຸ່ມການປິ່ນປົວທຽບກັບຈຸລັງໃນກຸ່ມຄວບຄຸມ,
ວິທີການຫຼາຍຄັ້ງທີ່ mRNA ຂອງ X ມີການປ່ຽນແປງ, ນີ້ແມ່ນປະລິມານທີ່ກ່ຽວຂ້ອງ;ໃນຈໍານວນຈຸລັງທີ່ແນ່ນອນ, mRNA ຂອງ X gene
ມີຈໍານວນສໍາເນົາຫຼາຍປານໃດ, ນີ້ແມ່ນປະລິມານຢ່າງແທ້ຈິງ.ປົກກະຕິແລ້ວສິ່ງທີ່ພວກເຮົາໃຊ້ຫຼາຍທີ່ສຸດໃນຫ້ອງທົດລອງແມ່ນວິທີການປະລິມານທີ່ກ່ຽວຂ້ອງ.ປົກກະຕິແລ້ວ,ວິທີການ 2-ΔΔctຖືກນໍາໃຊ້ຫຼາຍທີ່ສຸດໃນການທົດລອງ, ດັ່ງນັ້ນພຽງແຕ່ວິທີການນີ້ຈະຖືກນໍາສະເຫນີໃນລາຍລະອຽດທີ່ນີ້.
ວິທີການ 2-ΔΔct: ຜົນໄດ້ຮັບທີ່ໄດ້ຮັບແມ່ນຄວາມແຕກຕ່າງຂອງການສະແດງອອກຂອງ gene ເປົ້າຫມາຍໃນກຸ່ມທົດລອງທີ່ກ່ຽວຂ້ອງກັບ gene ເປົ້າຫມາຍໃນກຸ່ມຄວບຄຸມ.ມັນຈໍາເປັນຕ້ອງໃຫ້ປະສິດທິພາບການຂະຫຍາຍຂອງທັງ gene ເປົ້າຫມາຍແລະ gene ອ້າງອີງພາຍໃນແມ່ນຢູ່ໃກ້ກັບ 100%, ແລະ deviation ພີ່ນ້ອງບໍ່ຄວນເກີນ 5%.
ວິທີການຄິດໄລ່ມີດັ່ງນີ້:
ກຸ່ມຄວບຄຸມ Δct = ມູນຄ່າ ct ຂອງ gene ເປົ້າຫມາຍໃນກຸ່ມຄວບຄຸມ - ຄ່າ ct ຂອງ gene ອ້າງອີງພາຍໃນໃນກຸ່ມຄວບຄຸມ
ກຸ່ມທົດລອງ Δct = ຄ່າ ct ຂອງ gene ເປົ້າໝາຍໃນກຸ່ມທົດລອງ - ຄ່າ ct ຂອງ gene ອ້າງອີງພາຍໃນໃນກຸ່ມທົດລອງ
ΔΔct=Δct ກຸ່ມທົດລອງ-Δct ກຸ່ມຄວບຄຸມ
ສຸດທ້າຍ, ຄິດໄລ່ຄວາມຫຼາກຫຼາຍຂອງຄວາມແຕກຕ່າງໃນລະດັບການສະແດງອອກ:
ປ່ຽນ Fold = 2-Δ ct (ກົງກັນກັບຟັງຊັນ excel ແມ່ນ POWER)
ຜະລິດຕະພັນທີ່ກ່ຽວຂ້ອງ:
ເວລາປະກາດ: ພຶດສະພາ-20-2023