Ⅰ. ເພີ່ມຄວາມອ່ອນໄຫວຂອງລະບົບຕິກິຣິຍາ:
1. ແຍກ RNA ຄຸນະພາບສູງ:
ການສັງເຄາະ cDNA ທີ່ປະສົບຜົນສໍາເລັດແມ່ນມາຈາກ RNA ທີ່ມີຄຸນນະພາບສູງ.RNA ທີ່ມີຄຸນນະພາບສູງຄວນຮັບປະກັນຢ່າງຫນ້ອຍທັງຫມົດຍາວກວ່າແລະບໍ່ມີສານຍັບຍັ້ງທີ່ບໍ່ມີເອນໄຊການບັນທຶກ, ເຊັ່ນ EDTA ຫຼື SDS.ຄຸນນະພາບຂອງ RNA ກໍານົດມູນຄ່າສູງສຸດຂອງຂໍ້ມູນລໍາດັບທີ່ທ່ານສາມາດຖ່າຍທອດໄປຫາ cDNA.ວິທີການເຮັດຄວາມສະອາດ RNA ທົ່ວໄປແມ່ນວິທີການຂັ້ນຕອນຂອງການໃຊ້ isoocyanate/acidophenol.ເພື່ອປ້ອງກັນມົນລະພິດຂອງ RNase, RNA ທີ່ແຍກອອກຈາກຕົວຢ່າງທີ່ອຸດົມສົມບູນໃນ RNase (ເຊັ່ນ: pancreas) ຮຽກຮ້ອງໃຫ້ມີການເກັບຮັກສາ formaldehyde ເພື່ອຊ່ວຍປະຢັດ RNA ທີ່ມີຄຸນນະພາບສູງ, ເຊິ່ງຫຼາຍກວ່າເກົ່າສໍາລັບການເກັບຮັກສາໃນໄລຍະຍາວ.RNA ທີ່ສະກັດຈາກຕັບຫນູໄດ້ຖືກທໍາລາຍໂດຍພື້ນຖານແລ້ວຫຼັງຈາກຫນຶ່ງອາທິດຂອງການເກັບຮັກສາໃນນ້ໍາ, ໃນຂະນະທີ່ RNA ທີ່ສະກັດຈາກ spleen ຫນູຍັງຄົງຄົງທີ່ຫຼັງຈາກສາມປີເກັບຮັກສາໄວ້ໃນນ້ໍາ.ນອກຈາກນັ້ນ, ຂໍ້ຄວາມທີ່ໃຫຍ່ກວ່າ 4kb ແມ່ນມີຄວາມອ່ອນໄຫວຕໍ່ກັບການຕິດຕາມການເຊື່ອມໂຊມຂອງ RNase ຫຼາຍກວ່າການຖອດຂໍ້ຄວາມຂະໜາດນ້ອຍ.ເພື່ອເພີ່ມຄວາມຫມັ້ນຄົງຂອງຕົວຢ່າງ RNA ການເກັບຮັກສາ, RNA ສາມາດຖືກລະລາຍໃນ methalamine ຂອງ ion, ແລະເກັບຮັກສາໄວ້ -70 ° C.Thylide ທີ່ໃຊ້ເພື່ອຊ່ວຍປະຢັດ RNA ຈະຕ້ອງບໍ່ມີວັດຖຸອື່ນໆທີ່ທໍາລາຍ RNA.RNA, ທີ່ມາຈາກ pancreas, ສາມາດຖືກບັນທຶກໄວ້ໃນ methalmamine ຢ່າງຫນ້ອຍຫນຶ່ງປີ.ເມື່ອທ່ານພ້ອມທີ່ຈະໃຊ້ RNA, ທ່ານສາມາດນໍາໃຊ້ວິທີການດັ່ງຕໍ່ໄປນີ້ເພື່ອ precipitate RNA: ເພີ່ມ NaCl ກັບ 0.2m ແລະ 4 ເທົ່າຂອງປະລິມານຂອງເອທານອນ, ວາງອຸນຫະພູມຫ້ອງສໍາລັບ 3-5 ນາທີ, ແລະ 10,000 × g centrifugal ສໍາລັບ 5 ນາທີ.
2. ໃຊ້ reverse transcriptase ໂດຍບໍ່ມີກິດຈະກໍາ RNaseH (RNaseH-):
ຕົວຍັບຍັ້ງ RNase ມັກຈະຖືກເພີ່ມເຂົ້າໃນປະຕິກິລິຍາການຖອດຖອນຄືນໃຫມ່ເພື່ອເພີ່ມຄວາມຍາວແລະຜົນຜະລິດຂອງການສັງເຄາະ cDNA.RNase inhibitor ໄດ້ຖືກເພີ່ມເຂົ້າໃນປະຕິກິລິຍາການສັງເຄາະລະບົບຕ່ອງໂສ້ທໍາອິດໃນທີ່ປະທັບຂອງ buffers ແລະຕົວແທນການຫຼຸດຜ່ອນເຊັ່ນ DTT ເນື່ອງຈາກວ່າຂະບວນການສັງເຄາະກ່ອນ cDNA denatures inhibitor, ດັ່ງນັ້ນການປ່ອຍ RNases ຜູກມັດທີ່ degrade RNA.Protein RNase inhibitor ພຽງແຕ່ປ້ອງກັນການເຊື່ອມໂຊມຂອງ RNA ໂດຍ RNase A, B, C, ແລະບໍ່ປ້ອງກັນ RNases ເທິງຜິວຫນັງ, ດັ່ງນັ້ນການດູແລບໍ່ຄວນແນະນໍາ RNases ຈາກນິ້ວມືເຖິງວ່າຈະມີການໃຊ້ຢາຍັບຍັ້ງເຫຼົ່ານີ້.
Reverse transcriptase ກະຕຸ້ນການປ່ຽນ RNA ເປັນ cDNA.ທັງ M-MLV ແລະ AMV ມີກິດຈະກໍາ RNaseH endogenous ນອກເຫນືອໄປຈາກກິດຈະກໍາ polymerase ຂອງຕົນເອງ.ກິດຈະກໍາ RNaseH ແຂ່ງຂັນກັບກິດຈະກໍາ polymerase ສໍາລັບ strands heterozygous ສ້າງຕັ້ງຂຶ້ນລະຫວ່າງແມ່ແບບ RNA ແລະ primers DNA ຫຼືສາຍຂະຫຍາຍ cDNA, ແລະ degrades RNA: strands RNA ໃນສະລັບສັບຊ້ອນ DNA.ແມ່ແບບ RNA ທີ່ຊຸດໂຊມໂດຍກິດຈະກໍາ RNaseH ບໍ່ສາມາດຖືກນໍາໃຊ້ເປັນຊັ້ນຍ່ອຍທີ່ມີປະສິດທິພາບສໍາລັບການສັງເຄາະ cDNA, ການຫຼຸດຜ່ອນຜົນຜະລິດແລະຄວາມຍາວຂອງການສັງເຄາະ cDNA.ດັ່ງນັ້ນການກໍາຈັດຫຼືຫຼຸດລົງຢ່າງຫຼວງຫຼາຍກິດຈະກໍາ RNaseH ຂອງ reverse transcriptase ຈະເປັນປະໂຫຍດອັນໃຫຍ່ຫຼວງ.
SuperScriptⅡ reverse transcriptase, MMLV reverse transcriptase ຂອງ RNaseH- ແລະ thermoScript reverse transcriptase, AMV ຂອງ RNaseH- ໃຫ້ຜົນຜະລິດ cDNA ເຕັມຄວາມຍາວຫຼາຍກວ່າ MMLV ແລະ AMV.ຄວາມອ່ອນໄຫວ RT-PCR ໄດ້ຮັບຜົນກະທົບຈາກປະລິມານຂອງ cDNA ທີ່ສັງເຄາະ.ThermoScript ແມ່ນມີຄວາມອ່ອນໄຫວຫຼາຍກ່ວາ AMV.ຂະຫນາດຂອງຜະລິດຕະພັນ RT-PCR ແມ່ນຖືກຈໍາກັດໂດຍຄວາມສາມາດຂອງ reverse transcriptase ເພື່ອສັງເຄາະ cDNA, ໂດຍສະເພາະໃນເວລາທີ່ cloning Cdnas ຂະຫນາດໃຫຍ່.ເມື່ອປຽບທຽບກັບ MMLV, SuperScripⅡເພີ່ມຜົນຜະລິດຂອງຜະລິດຕະພັນ RT-PCR ຍາວຢ່າງຫຼວງຫຼາຍ.RNaseH-'s reverse transcriptase ຍັງເພີ່ມຄວາມຫມັ້ນຄົງຂອງຄວາມຮ້ອນ, ດັ່ງນັ້ນປະຕິກິລິຍາສາມາດປະຕິບັດໄດ້ໃນອຸນຫະພູມສູງກວ່າປົກກະຕິຂອງ 37-42 ℃.ພາຍໃຕ້ເງື່ອນໄຂການສັງເຄາະທີ່ແນະນໍາ, primers oligo(dT) ແລະ 10μCi [alpha-p]dCTP ໄດ້ຖືກນໍາໃຊ້.ການຜະລິດທັງຫມົດຂອງລະບົບຕ່ອງໂສ້ທໍາອິດໄດ້ຖືກຄິດໄລ່ໂດຍໃຊ້ວິທີການ precipitation TCA.cDNA ຄວາມຍາວເຕັມໄດ້ຖືກວິເຄາະໂດຍໃຊ້ການກໍາຈັດເສັ້ນດ່າງທີ່ມີຂະຫນາດແລະນັບຢູ່ໃນ gel agarose ເປັນດ່າງ.
3. ເພີ່ມອຸນຫະພູມຮັກສາຄວາມຮ້ອນຂອງ reverse transcription:
ອຸນຫະພູມຖືສູງຂຶ້ນຊ່ວຍເປີດໂຄງສ້າງຮອງຂອງ RNA ແລະເພີ່ມຜົນຜະລິດຂອງປະຕິກິລິຍາ.ສໍາລັບແມ່ແບບ RNA ສ່ວນໃຫຍ່, ການຖື RNA ແລະ primer ຢູ່ທີ່ 65 ° C ໂດຍບໍ່ມີ buffer ຫຼືເກືອແລະຫຼັງຈາກນັ້ນເຮັດໃຫ້ພວກມັນເຢັນຢ່າງໄວວາໃນກ້ອນຈະກໍາຈັດໂຄງສ້າງຮອງສ່ວນໃຫຍ່ແລະອະນຸຍາດໃຫ້ primers ຜູກມັດ.ຢ່າງໃດກໍຕາມ, ບາງແມ່ແບບຍັງຄົງມີໂຄງສ້າງຮອງ, ເຖິງແມ່ນວ່າຫຼັງຈາກການທໍາລາຍຄວາມຮ້ອນ.ການຂະຫຍາຍແມ່ແບບທີ່ຍາກເຫຼົ່ານີ້ສາມາດປະຕິບັດໄດ້ໂດຍໃຊ້ ThermoScript reverse transcriptase ແລະໂດຍການວາງປະຕິກິລິຍາ reverse transcriptase ໃນອຸນຫະພູມທີ່ສູງຂຶ້ນເພື່ອປັບປຸງການຂະຫຍາຍ.ອຸນຫະພູມການຖືທີ່ສູງຂຶ້ນຍັງສາມາດເພີ່ມຄວາມສະເພາະ, ໂດຍສະເພາະໃນເວລາທີ່ການສັງເຄາະ cDNA ຖືກປະຕິບັດໂດຍໃຊ້ primers ສະເພາະພັນທຸກໍາ (GSPS) (ເບິ່ງບົດທີ 3).ຖ້າໃຊ້ GSP, ໃຫ້ແນ່ໃຈວ່າຄ່າ Tm ຂອງ primer ແມ່ນຄືກັນກັບອຸນຫະພູມທີ່ຄາດໄວ້.ຢ່າໃຊ້ oligo(dT) ແລະ primers ແບບສຸ່ມສູງກວ່າ 60 ℃.primers Random ຈໍາເປັນຕ້ອງໄດ້ຈັດຂຶ້ນຢູ່ທີ່ 25 ℃ສໍາລັບ 10 ນາທີກ່ອນທີ່ຈະເພີ່ມຂຶ້ນເຖິງ 60 ℃.ນອກເໜືອໄປຈາກການໃຊ້ອຸນຫະພູມການຖອດຂໍ້ຄວາມແບບປີ້ນກັນທີ່ສູງຂຶ້ນ, ຄວາມສະເພາະສາມາດປັບປຸງໄດ້ໂດຍການຖ່າຍທອດສ່ວນປະສົມຂອງ RNA/ primer ໂດຍກົງຈາກອຸນຫະພູມ denaturing 65 ℃ ໄປສູ່ອຸນຫະພູມການຖອດຂໍ້ຄວາມແບບປີ້ນກັບກັນ ແລະ ເພີ່ມການປະສົມປະຕິກິລິຍາ 2 × preheated (ການສັງເຄາະຄວາມຮ້ອນເລີ່ມຕົ້ນ cDNA).ວິທີການນີ້ຊ່ວຍປ້ອງກັນການຈັບຄູ່ຖານ intermolecular ທີ່ເກີດຂື້ນໃນອຸນຫະພູມຕ່ໍາ.ການໃຊ້ເຄື່ອງມື PCR ເຮັດໃຫ້ການປ່ຽນອຸນຫະພູມຫຼາຍອັນທີ່ຕ້ອງການສໍາລັບ RT-PCR ງ່າຍຂຶ້ນ.
ໂພລີເມີເຣສຄົງທີ່ຄວາມຮ້ອນ tth ເຮັດຫນ້າທີ່ເປັນ DNA polymerase ໃນທີ່ປະທັບຂອງ Mg2+ ແລະ RNA polymerase ໃນທີ່ປະທັບຂອງ Mn2+.ມັນສາມາດຮັກສາຄວາມຮ້ອນໄດ້ເຖິງ 65 ℃.ຢ່າງໃດກໍ່ຕາມ, ການມີ Mn2+ ໃນລະຫວ່າງການ PCR ຫຼຸດຜ່ອນຄວາມຊື່ສັດ, ເຊິ່ງເຮັດໃຫ້ Tth polymerase ຫນ້ອຍທີ່ເຫມາະສົມສໍາລັບການຂະຫຍາຍຄວາມແມ່ນຍໍາສູງ, ເຊັ່ນ cDNA cloning.ນອກຈາກນັ້ນ, Tth ມີປະສິດທິພາບຫນ້ອຍໃນການຖອດຂໍ້ຄວາມແບບປີ້ນກັບກັນ, ເຊິ່ງຊ່ວຍຫຼຸດຜ່ອນຄວາມອ່ອນໄຫວ, ແລະເນື່ອງຈາກ enzyme ດຽວສາມາດປະຕິບັດການຖອດຂໍ້ຄວາມແບບປີ້ນກັບກັນແລະ PCR, ປະຕິກິລິຍາຄວບຄຸມໂດຍບໍ່ມີການຖອດຂໍ້ຄວາມຍ້ອນກັບບໍ່ສາມາດຖືກນໍາໃຊ້ເພື່ອຈໍາແນກຜະລິດຕະພັນຂະຫຍາຍໃຫຍ່ຂື້ນຂອງ cDNA ຈາກ DNA ພັນທຸ ກຳ ທີ່ປົນເປື້ອນ.
4. ສິ່ງເສີມທີ່ສົ່ງເສີມການຖອດຂໍ້ຄວາມແບບຍ້ອນກັບ:
ການເພີ່ມເຕີມຂອງສານເຕີມແຕ່ງ, ລວມທັງ glycerin ແລະ DMSO, ກັບຕິກິຣິຍາສັງເຄາະລະບົບຕ່ອງໂສ້ທໍາອິດສາມາດຫຼຸດຜ່ອນຄວາມຫມັ້ນຄົງຂອງສາຍສອງຂອງອາຊິດ nucleic ແລະ unwind ໂຄງສ້າງຮອງ RNA.ສາມາດເພີ່ມໄດ້ເຖິງ 20% glycerin ຫຼື 10% DMSO ໂດຍບໍ່ມີຜົນກະທົບຕໍ່ກິດຈະກໍາຂອງ SuperScriptⅡ ຫຼື MMLV.AMV ຍັງສາມາດທົນທານຕໍ່ເຖິງ 20% glycerol ໂດຍບໍ່ມີການຫຼຸດຜ່ອນກິດຈະກໍາ.ເພື່ອເພີ່ມຄວາມອ່ອນໄຫວຂອງ RT-PCR ໃນ SuperScriptⅡ ຕິກິຣິຍາ reverse transcription, 10% glycerol ສາມາດເພີ່ມແລະ insulated ຢູ່ທີ່ 45 ℃.ຖ້າ 1/10 ຂອງຜະລິດຕະພັນປະຕິກິລິຍາ retrotranscription-reaction ໄດ້ຖືກເພີ່ມໃສ່ PCR, ຄວາມເຂັ້ມຂົ້ນຂອງ glycerol ໃນປະຕິກິລິຍາຂະຫຍາຍແມ່ນ 0.4%, ເຊິ່ງບໍ່ພຽງພໍທີ່ຈະຍັບຍັ້ງ PCR.
5. ການປະມວນຜົນ RNaseH:
ຄວາມອ່ອນໄຫວສາມາດປັບປຸງໄດ້ໂດຍການປິ່ນປົວປະຕິກິລິຍາການສັງເຄາະ cDNA ກັບ RNaseH ກ່ອນ PCR.ສໍາລັບບາງແມ່ແບບ, ມັນຄິດວ່າ RNA ໃນປະຕິກິລິຍາສັງເຄາະ cDNA ປ້ອງກັນການຜູກມັດຂອງຜະລິດຕະພັນຂະຫຍາຍໃຫຍ່ຂື້ນ, ໃນກໍລະນີນີ້ການປິ່ນປົວ RNaseH ສາມາດເພີ່ມຄວາມອ່ອນໄຫວ.ໂດຍທົ່ວໄປແລ້ວ, ການປິ່ນປົວ RNaseH ແມ່ນຕ້ອງການສໍາລັບການຂະຫຍາຍຂອງແມ່ແບບເປົ້າຫມາຍ cDNA ທີ່ມີຄວາມຍາວຂ້ອນຂ້າງຍາວ, ເຊັ່ນ: tuberous scherosisⅡ ທີ່ມີສໍາເນົາຕ່ໍາ.ສໍາລັບແມ່ແບບທີ່ມີຄວາມຫຍຸ້ງຍາກນີ້, RNaseH ໄດ້ປັບປຸງສັນຍານທີ່ສ້າງຂຶ້ນໂດຍ cDNA ທີ່ສັງເຄາະໂດຍ SuperScriptⅡຫຼື AMV.ສໍາລັບປະຕິກິລິຍາ RT-PCR ສ່ວນໃຫຍ່, ການປິ່ນປົວ RNaseH ແມ່ນທາງເລືອກເພາະວ່າຂັ້ນຕອນ 95 ℃ insulated PCR denaturation ໂດຍປົກກະຕິ hydrolyzes RNA ຈາກ RNA: DNA complex.
6. ປັບປຸງວິທີການກວດຫາ RNA ຂະໜາດນ້ອຍ:
RT-PCR ມີຄວາມທ້າທາຍໂດຍສະເພາະເມື່ອມີ RNA ຈໍານວນນ້ອຍໆເທົ່ານັ້ນ.ການເພີ່ມ glycogen ເປັນຜູ້ໃຫ້ບໍລິການໃນລະຫວ່າງການແຍກ RNA ຊ່ວຍເພີ່ມຜົນຜະລິດຂອງຕົວຢ່າງຂະຫນາດນ້ອຍ.ສາມາດເພີ່ມ glycogen ທີ່ບໍ່ມີ RNase ໃນເວລາດຽວກັນກັບ Trizol.Glycogen ແມ່ນລະລາຍໃນນ້ໍາແລະສາມາດຍັງຄົງຢູ່ໃນໄລຍະນ້ໍາທີ່ມີ RNA ເພື່ອຊ່ວຍໃນຝົນຕົກຕໍ່ມາ.ຄວາມເຂັ້ມຂຸ້ນຂອງ glycogen ທີ່ບໍ່ມີ RNase ທີ່ແນະນໍາແມ່ນ 250μg / ml ສໍາລັບຕົວຢ່າງຫນ້ອຍກວ່າ 50mg ຂອງເນື້ອເຍື່ອຫຼື 106 ຈຸລັງວັດທະນະທໍາ.
ການເພີ່ມ BSA acetylated ເພື່ອປະຕິກິລິຢາ reverse transcription ໂດຍໃຊ້ SuperScriptⅡສາມາດເພີ່ມຄວາມອ່ອນໄຫວ, ແລະສໍາລັບ RNA ຂະຫນາດນ້ອຍ, ການຫຼຸດຜ່ອນປະລິມານຂອງ SuperScriptⅡ ແລະການເພີ່ມ 40 ຫນ່ວຍຂອງ RnaseOut nuclease inhibitor ສາມາດປັບປຸງລະດັບການຊອກຄົ້ນຫາ.ຖ້າ glycogen ຖືກນໍາໃຊ້ໃນການແຍກ RNA, ການເພີ່ມ BSA ຫຼື RNase inhibitors ເພື່ອປະຕິກິລິຍາ reverse transcription ໂດຍໃຊ້ SuperScriptⅡແມ່ນຍັງແນະນໍາ.
Ⅱ. ເພີ່ມຄວາມສະເພາະຂອງ RT-PCR
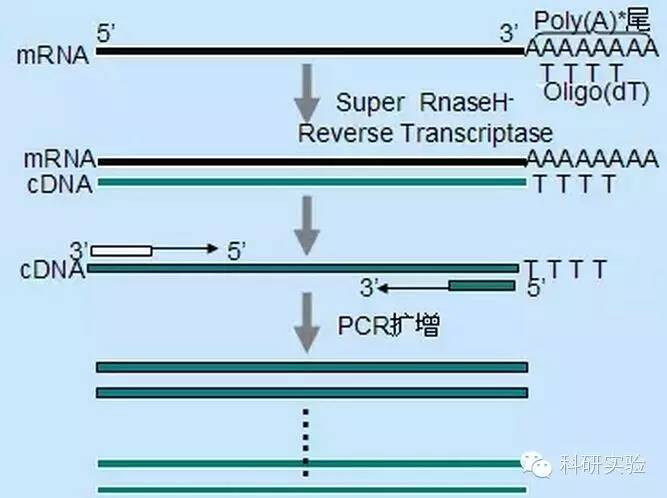
1. ການສັງເຄາະ cNDA:
ສາມວິທີທີ່ແຕກຕ່າງກັນສາມາດຖືກນໍາໃຊ້ເພື່ອລິເລີ່ມການສັງເຄາະ cDNA strand ທໍາອິດ, ແລະຄວາມສະເພາະຂອງແຕ່ລະວິທີການມີຜົນກະທົບຕໍ່ປະລິມານແລະປະເພດຂອງ cDNA ສັງເຄາະ.
ວິທີການ primer ແບບສຸ່ມແມ່ນສະເພາະຫນ້ອຍທີ່ສຸດໃນສາມວິທີການ.primers ໄດ້ຖືກ annealed ຢູ່ຫຼາຍສະຖານທີ່ຕະຫຼອດການຖອດຂໍ້ຄວາມເພື່ອຜະລິດ cDNA ສັ້ນ, ຄວາມຍາວບາງສ່ວນ.ວິທີການນີ້ມັກຈະຖືກນໍາໃຊ້ເພື່ອໃຫ້ໄດ້ 5′ ລໍາດັບ terminal ແລະ cDNA ຈາກແມ່ແບບ RNA ກັບພາກພື້ນໂຄງສ້າງຂັ້ນສອງຫຼືກັບສະຖານທີ່ສິ້ນສຸດທີ່ reverse transcriptase ບໍ່ສາມາດ replicate.ເພື່ອໃຫ້ໄດ້ cDNA ທີ່ຍາວທີ່ສຸດ, ອັດຕາສ່ວນຂອງ primers ກັບ RNA ໃນແຕ່ລະຕົວຢ່າງ RNA ຈໍາເປັນຕ້ອງໄດ້ຖືກກໍານົດໂດຍທາງກົງ.ຄວາມເຂັ້ມຂົ້ນເບື້ອງຕົ້ນຂອງ primers ແບບສຸ່ມແມ່ນຕັ້ງແຕ່ 50 ຫາ 250ng ຕໍ່ລະບົບປະຕິກິລິຍາ20μl.ເນື່ອງຈາກວ່າ cDNA ສັງເຄາະຈາກ RNA ທັງຫມົດໂດຍໃຊ້ primers random ສ່ວນໃຫຍ່ແມ່ນ RNA ribosomal, poly(A) + RNA ໂດຍທົ່ວໄປແມ່ນເລືອກເປັນແມ່ແບບ.
ການລິເລີ່ມ Oligo(dT) ແມ່ນສະເພາະຫຼາຍກ່ວາ primers ແບບສຸ່ມ.ມັນປະສົມກັບຫາງ poly(A) ທີ່ພົບເຫັນຢູ່ປາຍ 3′ ຂອງ mRNA ໃນຈຸລັງ eukaryotic ສ່ວນໃຫຍ່.ເນື່ອງຈາກວ່າ poly(A) + RNA ແມ່ນປະມານ 1% ຫາ 2% ຂອງ RNA ທັງຫມົດ, ຈໍານວນແລະຄວາມຊັບຊ້ອນຂອງ cDNA ແມ່ນຫນ້ອຍກວ່າຖ້າໃຊ້ primers ແບບສຸ່ມ.ເນື່ອງຈາກຄວາມສະເພາະສູງຂອງມັນ, oligo(dT) ໂດຍທົ່ວໄປແລ້ວບໍ່ຮຽກຮ້ອງໃຫ້ມີການເພີ່ມປະສິດທິພາບສໍາລັບ RNA ກັບອັດຕາສ່ວນ primer ແລະການເລືອກ poly(A)+.ແນະນໍາໃຫ້ໃຊ້ 0.5μg oligo(dT) ຕໍ່ລະບົບຕິກິຣິຍາ20μl.oligo(dT)12-18 ແມ່ນເຫມາະສົມສໍາລັບ RT-PCR ຫຼາຍທີ່ສຸດ.ThermoScript RT-PCR System ສະຫນອງ oligo(dT)20 ເນື່ອງຈາກຄວາມຫມັ້ນຄົງຂອງຄວາມຮ້ອນທີ່ດີແລະເຫມາະສົມສໍາລັບອຸນຫະພູມຖືທີ່ສູງຂຶ້ນ.
primers ສະເພາະພັນທຸກໍາ (GSP) ແມ່ນ primers ສະເພາະທີ່ດີທີ່ສຸດສໍາລັບຂັ້ນຕອນການຖອດຂໍ້ຄວາມແບບປີ້ນກັບກັນ.GSP ເປັນ oligonucleoside antisense ທີ່ສາມາດປະສົມໂດຍສະເພາະກັບລໍາດັບປາຍທາງ RNA, ແທນທີ່ຈະເຮັດໃຫ້ Rnas ທັງຫມົດເຊັ່ນ primers random ຫຼື oligo (dT).ກົດລະບຽບທີ່ໃຊ້ໃນການອອກແບບ primers PCR ຍັງໃຊ້ກັບການອອກແບບປະຕິກິລິຍາ reverse transcription GSP.GSP ສາມາດເປັນລໍາດັບດຽວກັນກັບ primer amplification annealed ໃນຕອນທ້າຍຂອງ mRNA3′, ຫຼື GSP ສາມາດຖືກອອກແບບເພື່ອ annealed ລົງນ້ໍາກັບ primer ຂະຫຍາຍ reverse.ສໍາລັບວັດຖຸຂະຫຍາຍບາງອັນ, ມັນຈໍາເປັນຕ້ອງອອກແບບ primer antisense ຫຼາຍກວ່າຫນຶ່ງສໍາລັບ RT-PCR ສົບຜົນສໍາເລັດເພາະວ່າໂຄງສ້າງຂັ້ນສອງຂອງ RNA ເປົ້າຫມາຍອາດຈະປ້ອງກັນບໍ່ໃຫ້ primer ຜູກມັດ.ມັນໄດ້ຖືກແນະນໍາໃຫ້ໃຊ້ 1pmol antisense GSP ໃນລະບົບຕິກິຣິຍາສັງເຄາະລະບົບຕ່ອງໂສ້ທໍາອິດຂອງ20μl.
2. ເພີ່ມອຸນຫະພູມຮັກສາຄວາມຮ້ອນຂອງ reverse transcription:
ເພື່ອໃຊ້ປະໂຫຍດອັນເຕັມທີ່ຂອງສະເພາະ GSP, reverse transcriptase ທີ່ມີຄວາມຫມັ້ນຄົງຄວາມຮ້ອນສູງຄວນຖືກນໍາໃຊ້.ຄວາມຮ້ອນທີ່ຄົງທີ່ transcriptase ສາມາດຖືກ insulated ໃນອຸນຫະພູມທີ່ສູງຂຶ້ນເພື່ອເພີ່ມຄວາມເຂັ້ມງວດຕິກິຣິຍາ.ຕົວຢ່າງ, ຖ້າ GSP ຖືກ annealed ທີ່ 55 ° C, ຫຼັງຈາກນັ້ນ, ສະເພາະຂອງ GSP ບໍ່ໄດ້ຖືກນໍາໃຊ້ຢ່າງເຕັມທີ່ຖ້າຫາກວ່າການຖອດຂໍ້ຄວາມແບບປີ້ນກັບກັນໄດ້ຖືກປະຕິບັດຢູ່ທີ່ 37 ° C ດ້ວຍຄວາມເຄັ່ງຄັດຕ່ໍາໂດຍໃຊ້ AMV ຫຼື M-MLV.ຢ່າງໃດກໍຕາມ, SuperScripⅡແລະ ThermoScript ສາມາດປະຕິກິລິຍາຢູ່ທີ່ 50 ℃ຫຼືສູງກວ່າ, ເຊິ່ງກໍາຈັດຜະລິດຕະພັນທີ່ບໍ່ສະເພາະທີ່ຜະລິດຢູ່ໃນອຸນຫະພູມຕ່ໍາ.ສໍາລັບຄວາມສະເພາະສູງສຸດ, ການປະສົມ RNA / primer ສາມາດຖືກໂອນໂດຍກົງຈາກອຸນຫະພູມ denaturation 65 ℃ໄປຫາອຸນຫະພູມການຖອດຂໍ້ຄວາມແບບປີ້ນກັບກັນດ້ວຍການເພີ່ມຂອງປະສົມປະຕິກິລິຍາ 2 x preheated (ການເລີ່ມຕົ້ນຄວາມຮ້ອນຂອງການສັງເຄາະ cDNA).ນີ້ຊ່ວຍປ້ອງກັນການຈັບຄູ່ພື້ນຖານລະຫວ່າງໂມເລກຸນຢູ່ໃນອຸນຫະພູມຕ່ໍາ.ການນໍາໃຊ້ເຄື່ອງມື PCR ເປັນຄວາມງ່າຍຂອງການປ່ຽນແປງອຸນຫະພູມຈໍານວນຫຼາຍທີ່ຈໍາເປັນສໍາລັບ RT-PCR.
3. ຫຼຸດຜ່ອນການປົນເປື້ອນ DNA ພັນທຸ ກຳ:
ຄວາມຫຍຸ້ງຍາກທີ່ເປັນໄປໄດ້ຫນຶ່ງກັບ RT-PCR ແມ່ນວ່າ RNA ປົນເປື້ອນ DNA ພັນທຸ ກຳ.ການນໍາໃຊ້ວິທີການແຍກ RNA ທີ່ດີກວ່າ, ເຊັ່ນ Trizol Reagent, ຫຼຸດຜ່ອນການປົນເປື້ອນ DNA genomic ໃນການກະກຽມ RNA.ເພື່ອຫຼີກເວັ້ນຜະລິດຕະພັນທີ່ຜະລິດຈາກ DNA genomic, RNA ສາມາດຖືກປະຕິບັດດ້ວຍການຂະຫຍາຍລະດັບ DnasⅠ ເພື່ອເອົາ DNA ທີ່ປົນເປື້ອນອອກກ່ອນການຖອດຂໍ້ຄວາມຄືນ.ຕົວຢ່າງໄດ້ຖືກເກັບຮັກສາໄວ້ຢູ່ທີ່ 65 ℃ໃນ 2.0mM EDTA ເປັນເວລາ 10 ນາທີເພື່ອຢຸດການຍ່ອຍອາຫານ DNaseⅠ.EDTA chelates magnesium ions ເພື່ອປ້ອງກັນບໍ່ໃຫ້ magnesium ion dependent RNA hydrolysis ທີ່ເກີດຂື້ນໃນອຸນຫະພູມສູງ.
ເພື່ອແຍກ cDNA ຂະຫຍາຍອອກຈາກຜະລິດຕະພັນຂະຫຍາຍ DNA ຂອງ genome, primers ທີ່ anneal ແຍກຕ່າງຫາກກັບ exon ແຍກສາມາດໄດ້ຮັບການອອກແບບ.ຜະລິດຕະພັນ PCR ທີ່ມາຈາກ cDNA ຈະສັ້ນກວ່າຜະລິດຕະພັນທີ່ມາຈາກ DNA ພັນທຸ ກຳ ທີ່ປົນເປື້ອນ.ການທົດລອງທີ່ຄວບຄຸມໂດຍບໍ່ມີການຖອດຖອນຄືນຂໍ້ມູນຍັງຖືກປະຕິບັດຢູ່ໃນແຕ່ລະແມ່ແບບ RNA ເພື່ອກໍານົດວ່າຊິ້ນທີ່ໄດ້ຮັບແມ່ນມາຈາກ DNA ພັນທຸກໍາຫຼື cDNA.ຜະລິດຕະພັນ PCR ທີ່ໄດ້ຮັບໃນກໍລະນີທີ່ບໍ່ມີການຖອດຖອນຄືນແມ່ນມາຈາກ genome.
ຜະລິດຕະພັນທີ່ກ່ຽວຂ້ອງ
RT-PCR ງ່າຍᵀᴹຂ້ອຍ (ຂັ້ນຕອນດຽວ)
- ຊຸດຫນຶ່ງຂັ້ນຕອນເຮັດໃຫ້ການຖອດຂໍ້ຄວາມແບບປີ້ນກັບກັນແລະ PCR ດໍາເນີນການຢູ່ໃນທໍ່ດຽວກັນ.ມັນພຽງແຕ່ຕ້ອງການເພີ່ມແມ່ແບບ RNA, primers PCR ສະເພາະແລະ RNase-Free ddH2O.
- ການວິເຄາະປະລິມານໃນເວລາທີ່ແທ້ຈິງຂອງ RNA ສາມາດໄດ້ຮັບການດໍາເນີນການຢ່າງວ່ອງໄວແລະຖືກຕ້ອງ.
-ຊຸດດັ່ງກ່າວໃຊ້ reagent Reverse transcription Foregene ທີ່ເປັນເອກະລັກ ແລະ Foregene HotStar Taq DNA Polymerase ປະສົມປະສານກັບລະບົບຕິກິຣິຍາທີ່ເປັນເອກະລັກເພື່ອປັບປຸງປະສິດທິພາບການຂະຫຍາຍ ແລະ ສະເພາະຂອງຕິກິຣິຍາ.
- ລະບົບຕິກິຣິຍາທີ່ດີທີ່ສຸດເຮັດໃຫ້ປະຕິກິລິຍາມີຄວາມອ່ອນໄຫວໃນການກວດສອບສູງ, ຄວາມຫມັ້ນຄົງຂອງຄວາມຮ້ອນທີ່ເຂັ້ມແຂງ, ແລະຄວາມທົນທານທີ່ດີກວ່າ.
- ຄວາມສາມາດໃນການກໍາຈັດ gDNA ທີ່ມີປະສິດທິພາບ, ເຊິ່ງສາມາດເອົາ gDNA ອອກໃນແມ່ແບບພາຍໃນ 2 ນາທີ.
- ລະບົບການຖອດຂໍ້ຄວາມແບບປີ້ນກັບກັນທີ່ມີປະສິດທິພາບ, ມັນໃຊ້ເວລາພຽງແຕ່ 15 ນາທີເພື່ອສໍາເລັດການສັງເຄາະ cDNA strand ທໍາອິດ.
- ແມ່ແບບທີ່ຊັບຊ້ອນ: ແມ່ແບບທີ່ມີເນື້ອໃນ GC ສູງແລະໂຄງສ້າງມັດທະຍົມທີ່ສັບສົນຍັງສາມາດຖືກຖອນຄືນດ້ວຍປະສິດທິພາບສູງ.
- ລະບົບການຖອດຂໍ້ຄວາມແບບປີ້ນກັບຄວາມອ່ອນໄຫວສູງ, ແມ່ແບບລະດັບ pg ສາມາດໄດ້ຮັບ cDNA ທີ່ມີຄຸນນະພາບສູງ.
- ລະບົບການຖອດຖອນຄືນໄດ້ມີຄວາມຫມັ້ນຄົງຄວາມຮ້ອນສູງ, ອຸນຫະພູມຕິກິຣິຍາທີ່ດີທີ່ສຸດແມ່ນ 42 ℃, ແລະມັນຍັງມີປະສິດທິພາບ reverse transcription ທີ່ດີທີ່ 50 ℃.
ເວລາປະກາດ: 07-07-2023