一, ເພີ່ມຄວາມອ່ອນໄຫວຂອງລະບົບຕິກິຣິຍາ:
1. ແຍກ RNA ຄຸນນະພາບສູງ:
ການສັງເຄາະ cDNA ທີ່ປະສົບຜົນສໍາເລັດແມ່ນມາຈາກ RNA ທີ່ມີຄຸນນະພາບສູງ.RNA ທີ່ມີຄຸນນະພາບສູງຢ່າງຫນ້ອຍຄວນຈະມີຄວາມຍາວເຕັມແລະບໍ່ມີຕົວຍັບຍັ້ງ transcriptase ແບບປີ້ນກັບກັນເຊັ່ນ EDTA ຫຼື SDS.ຄຸນນະພາບຂອງ RNA ກໍານົດຈໍານວນສູງສຸດຂອງຂໍ້ມູນລໍາດັບທີ່ທ່ານສາມາດຖ່າຍທອດເຂົ້າໄປໃນ cDNA.ວິທີການເຮັດຄວາມສະອາດ RNA ທົ່ວໄປແມ່ນວິທີການຫນຶ່ງຂັ້ນຕອນໂດຍໃຊ້ guanidine isothiocyanate / acid phenol.ເພື່ອປ້ອງກັນການປົນເປື້ອນດ້ວຍປະລິມານ RNase, RNA ທີ່ແຍກອອກຈາກຕົວຢ່າງທີ່ອຸດົມສົມບູນ RNase (ເຊັ່ນ: pancreas) ຕ້ອງໄດ້ຮັບການເກັບຮັກສາໄວ້ໃນ formaldehyde ເພື່ອຮັກສາ RNA ທີ່ມີຄຸນນະພາບສູງ, ໂດຍສະເພາະສໍາລັບການເກັບຮັກສາໃນໄລຍະຍາວ.RNA ທີ່ສະກັດຈາກຕັບຫນູໄດ້ຖືກທໍາລາຍໂດຍພື້ນຖານແລ້ວຫຼັງຈາກຖືກເກັບໄວ້ໃນນ້ໍາຫນຶ່ງອາທິດ, ໃນຂະນະທີ່ RNA ທີ່ສະກັດຈາກ spleen ຫນູຍັງຄົງຄົງທີ່ຫຼັງຈາກຖືກເກັບໄວ້ໃນນ້ໍາເປັນເວລາ 3 ປີ.ນອກຈາກນັ້ນ, ຂໍ້ຄວາມທີ່ຍາວກວ່າ 4 kb ແມ່ນມີຄວາມອ່ອນໄຫວຕໍ່ກັບການຍ່ອຍສະຫຼາຍໂດຍການຕິດຕາມ RNases ຫຼາຍກ່ວາການຖອດຂໍ້ຄວາມຂະຫນາດນ້ອຍ.ເພື່ອເພີ່ມຄວາມຫມັ້ນຄົງຂອງຕົວຢ່າງ RNA ທີ່ເກັບຮັກສາໄວ້, RNA ສາມາດຖືກລະລາຍໃນ deionized formamide ແລະເກັບຮັກສາໄວ້ທີ່ -70 ° C.Formamide ທີ່ໃຊ້ເພື່ອຮັກສາ RNA ຈະຕ້ອງບໍ່ມີສິ່ງເສດເຫຼືອທີ່ທໍາລາຍ RNA.RNA ຈາກ pancreas ສາມາດຮັກສາໄວ້ຢູ່ໃນ formamide ຢ່າງຫນ້ອຍຫນຶ່ງປີ.ໃນເວລາທີ່ກະກຽມການນໍາໃຊ້ RNA, ທ່ານສາມາດນໍາໃຊ້ວິທີດັ່ງຕໍ່ໄປນີ້ເພື່ອ precipitate RNA: ເພີ່ມ NaCl ເປັນ 0.2M ແລະ 4 ເທົ່າຂອງປະລິມານຂອງເອທານອນ, ວາງໄວ້ທີ່ອຸນຫະພູມຫ້ອງສໍາລັບ 3-5 ນາທີ, ແລະ centrifuge 10,000 × g ສໍາລັບ 5 ນາທີ.
2. ໃຊ້ RNaseH-inactive (RNaseH-) reverse transcriptase:
ຕົວຍັບຍັ້ງ RNase ມັກຈະຖືກເພີ່ມເຂົ້າໃນປະຕິກິລິຍາການຖອດຖອນຄືນໃຫມ່ເພື່ອເພີ່ມຄວາມຍາວແລະຜົນຜະລິດຂອງການສັງເຄາະ cDNA.ຢາຍັບຍັ້ງ RNase ຄວນໄດ້ຮັບການເພີ່ມໃນລະຫວ່າງປະຕິກິລິຍາສັງເຄາະສາຍທໍາອິດໃນທີ່ປະທັບຂອງ buffer ແລະຕົວຫຼຸດຜ່ອນ (ເຊັ່ນ: DTT), ເພາະວ່າຂະບວນການກ່ອນການສັງເຄາະ cDNA ເປັນຕົວຍັບຍັ້ງ, ດັ່ງນັ້ນຈຶ່ງປ່ອຍ RNase ຜູກມັດທີ່ສາມາດທໍາລາຍ RNA.Protein RNase inhibitors ພຽງແຕ່ປ້ອງກັນການເຊື່ອມໂຊມຂອງ RNA ໂດຍ RNase A, B, C, ແລະບໍ່ປ້ອງກັນ RNase ເທິງຜິວຫນັງ, ດັ່ງນັ້ນຈົ່ງລະມັດລະວັງບໍ່ໃຫ້ແນະນໍາ RNase ຈາກນິ້ວມືຂອງທ່ານເຖິງວ່າຈະມີການໃຊ້ inhibitors ເຫຼົ່ານີ້.
Reverse transcriptase ກະຕຸ້ນການປ່ຽນ RNA ເປັນ cDNA.ທັງ M-MLV ແລະ AMV ມີກິດຈະກໍາ RNaseH endogenous ນອກເຫນືອໄປຈາກກິດຈະກໍາ polymerase ຂອງຕົນເອງ.ກິດຈະກໍາ RNaseH ແລະກິດຈະກໍາ polymerase ແຂ່ງຂັນກັນສໍາລັບສາຍພັນປະສົມທີ່ສ້າງຂຶ້ນລະຫວ່າງແມ່ແບບ RNA ແລະສາຍພັນ DNA primer ຫຼື cDNA ຂະຫຍາຍ, ແລະເຮັດໃຫ້ສາຍພັນ RNA ຫຼຸດລົງໃນສະລັບສັບຊ້ອນ RNA: DNA.ແມ່ແບບ RNA ທີ່ເສື່ອມໂຊມໂດຍກິດຈະກໍາ RNaseH ບໍ່ສາມາດຮັບໃຊ້ເປັນຊັ້ນຍ່ອຍທີ່ມີປະສິດທິພາບສໍາລັບການສັງເຄາະ cDNA, ເຊິ່ງຫຼຸດລົງຜົນຜະລິດແລະຄວາມຍາວຂອງການສັງເຄາະ cDNA.ດັ່ງນັ້ນ, ມັນຈະເປັນປະໂຫຍດທີ່ຈະກໍາຈັດຫຼືຫຼຸດລົງຢ່າງຫຼວງຫຼາຍກິດຈະກໍາ RNaseH ຂອງ reverse transcriptase..
SuperScript Ⅱ reverse transcriptase, RNaseH- MMLV reverse transcriptase ແລະ thermoScript reverse transcriptase, RNaseH- AMV, ສາມາດໄດ້ຮັບປະລິມານຫຼາຍແລະ cDNA ເຕັມຄວາມຍາວຫຼາຍກ່ວາ MMLV ແລະ AMV.ຄວາມອ່ອນໄຫວ RT-PCR ຈະໄດ້ຮັບຜົນກະທົບຈາກປະລິມານການສັງເຄາະ cDNA.ThermoScript ແມ່ນມີຄວາມອ່ອນໄຫວຫຼາຍກ່ວາ AMV.ຂະຫນາດຂອງຜະລິດຕະພັນ RT-PCR ແມ່ນຖືກຈໍາກັດໂດຍຄວາມສາມາດຂອງ reverse transcriptase ເພື່ອສັງເຄາະ cDNA, ໂດຍສະເພາະໃນເວລາທີ່ cloning cDNAs ຂະຫນາດໃຫຍ່.ເມື່ອປຽບທຽບກັບ MMLV, SuperScripⅡເພີ່ມຜົນຜະລິດຂອງຜະລິດຕະພັນ RT-PCR ຍາວຢ່າງຫຼວງຫຼາຍ.RNaseH-reverse transcriptase ຍັງມີຄວາມທົນທານຕໍ່ຄວາມຮ້ອນເພີ່ມຂຶ້ນ, ດັ່ງນັ້ນປະຕິກິລິຍາສາມາດປະຕິບັດໄດ້ໃນອຸນຫະພູມທີ່ສູງກວ່າປົກກະຕິ 37-42 ° C.ພາຍໃຕ້ເງື່ອນໄຂການສັງເຄາະທີ່ແນະນໍາ, ໃຫ້ໃຊ້ primer oligo(dT) ແລະ 10 μCi ຂອງ [α-P]dCTP.ຜົນຜະລິດທັງຫມົດຂອງສາຍທໍາອິດໄດ້ຖືກຄິດໄລ່ໂດຍນໍາໃຊ້ວິທີການ precipitation TCA.cDNA ຄວາມຍາວເຕັມໄດ້ຖືກວິເຄາະໂດຍໃຊ້ແຖບຈັດຮຽງຂະຫນາດແລະນັບຢູ່ໃນ gel agarose alkaline.
3. ເພີ່ມອຸນຫະພູມ incubation ສໍາລັບ reverse transcription:
ອຸນຫະພູມ incubation ສູງຂຶ້ນຊ່ວຍເປີດໂຄງສ້າງຮອງ RNA, ເພີ່ມຜົນຜະລິດຂອງຕິກິຣິຍາ.ສໍາລັບແມ່ແບບ RNA ສ່ວນໃຫຍ່, ການ incubating RNA ແລະ primers ຢູ່ທີ່ 65 ° C ໂດຍບໍ່ມີການ buffer ຫຼືເກືອ, ປະຕິບັດຕາມໂດຍການເຢັນຢ່າງໄວວາກ່ຽວກັບກ້ອນຈະກໍາຈັດໂຄງສ້າງຮອງສ່ວນໃຫຍ່ແລະອະນຸຍາດໃຫ້ primers ຜູກມັດ.ຢ່າງໃດກໍຕາມ, ບາງແມ່ແບບຍັງຄົງມີໂຄງສ້າງຮອງ, ເຖິງແມ່ນວ່າຫຼັງຈາກການທໍາລາຍຄວາມຮ້ອນ.ການຂະຫຍາຍຕົວແບບທີ່ຍາກເຫຼົ່ານີ້ສາມາດປະຕິບັດໄດ້ໂດຍໃຊ້ ThermoScript Reverse Transcriptase ແລະວາງປະຕິກິລິຍາການຖອດຂໍ້ຄວາມແບບປີ້ນກັບກັນຢູ່ໃນອຸນຫະພູມທີ່ສູງຂຶ້ນເພື່ອປັບປຸງການຂະຫຍາຍ.ອຸນຫະພູມ incubation ສູງຂຶ້ນຍັງສາມາດເພີ່ມຄວາມສະເພາະ, ໂດຍສະເພາະໃນເວລາທີ່ primers gene-specific primers (GSP) ຖືກນໍາໃຊ້ສໍາລັບການສັງເຄາະ cDNA (ເບິ່ງບົດທີ 3).ຖ້າໃຊ້ GSP, ໃຫ້ແນ່ໃຈວ່າ Tm ຂອງ primers ແມ່ນຄືກັນກັບອຸນຫະພູມ incubation ທີ່ຄາດໄວ້.ຢ່າໃຊ້ oligo(dT) ແລະ primers ແບບສຸ່ມສູງກວ່າ 60 ° C.primers ແບບສຸ່ມຕ້ອງການ incubation ຢູ່ທີ່ 25 ° C ສໍາລັບ 10 ນາທີກ່ອນທີ່ຈະເພີ່ມຂຶ້ນເຖິງ 60 ° C.ນອກເໜືອໄປຈາກການໃຊ້ອຸນຫະພູມການຖອດຂໍ້ຄວາມແບບປີ້ນກັບກັນທີ່ສູງຂຶ້ນ, ຄວາມສະເພາະຂອງຍັງສາມາດປັບປຸງໄດ້ໂດຍການຖ່າຍທອດ RNA/primer mix ໂດຍກົງຈາກອຸນຫະພູມ denaturation 65°C ໄປສູ່ອຸນຫະພູມ incubation transcription reverse ແລະເພີ່ມປະສົມປະຕິກິລິຢາ 2× ທີ່ອົບອຸ່ນກ່ອນ (cDNA hot-start synthesis).ວິທີການນີ້ຊ່ວຍປ້ອງກັນການຈັບຄູ່ຖານ intermolecular ທີ່ເກີດຂື້ນໃນອຸນຫະພູມຕ່ໍາ.ການປ່ຽນອຸນຫະພູມຫຼາຍອັນທີ່ຕ້ອງການສໍາລັບ RT-PCR ສາມາດງ່າຍດາຍໄດ້ໂດຍການໃຊ້ເຄື່ອງສູບຄວາມຮ້ອນ.
Tth thermostable polymerase ເຮັດຫນ້າທີ່ເປັນ DNA polymerase ໃນທີ່ປະທັບຂອງ Mg2+ ແລະເປັນ RNA polymerase ໃນທີ່ປະທັບຂອງ Mn2+.ມັນສາມາດຮັກສາຄວາມອົບອຸ່ນຢູ່ທີ່ອຸນຫະພູມສູງສຸດຂອງ 65 ° C.ຢ່າງໃດກໍ່ຕາມ, ການປະກົດຕົວຂອງ Mn2+ ໃນລະຫວ່າງການ PCR ຫຼຸດຜ່ອນຄວາມຊື່ສັດ, ເຊິ່ງເຮັດໃຫ້ Tth polymerase ຫນ້ອຍທີ່ເຫມາະສົມສໍາລັບການຂະຫຍາຍຄວາມຊັດເຈນສູງ, ເຊັ່ນ: ການໂຄນຂອງ cDNA.ນອກຈາກນັ້ນ, Tth ມີປະສິດທິພາບ transcription ຕ່ໍາ, ເຊິ່ງຫຼຸດຜ່ອນຄວາມອ່ອນໄຫວ, ແລະ, ນັບຕັ້ງແຕ່ transcription reverse ແລະ PCR ສາມາດປະຕິບັດດ້ວຍ enzyme ດຽວ, ປະຕິກິລິຍາຄວບຄຸມໂດຍບໍ່ມີການ reverse transcription ບໍ່ສາມາດຖືກນໍາໃຊ້ເພື່ອປຽບທຽບຜະລິດຕະພັນຂະຫຍາຍ cDNA ກັບ DNA genomic ປົນເປື້ອນ.ຜະລິດຕະພັນຂະຫຍາຍໃຫຍ່ໄດ້ແຍກອອກ.
4. ສານເສີມທີ່ສົ່ງເສີມການຖອດຂໍ້ຄວາມແບບຍ້ອນກັບ:
ສານເຕີມແຕ່ງລວມທັງ glycerol ແລະ DMSO ໄດ້ຖືກເພີ່ມເຂົ້າໃນປະຕິກິລິຍາສັງເຄາະສາຍທໍາອິດ, ເຊິ່ງສາມາດຫຼຸດຜ່ອນຄວາມຫມັ້ນຄົງຂອງອາຊິດນິວຄລີອິກສອງສາຍແລະແກ້ໄຂໂຄງສ້າງຂັ້ນສອງຂອງ RNA.ສາມາດເພີ່ມໄດ້ເຖິງ 20% glycerol ຫຼື 10% DMSO ໂດຍບໍ່ມີຜົນກະທົບຕໍ່ກິດຈະກໍາ SuperScript II ຫຼື MMLV.AMV ຍັງສາມາດທົນທານຕໍ່ເຖິງ 20% glycerol ໂດຍບໍ່ມີການສູນເສຍກິດຈະກໍາ.ເພື່ອເພີ່ມຄວາມອ່ອນໄຫວຂອງ RT-PCR ໃນປະຕິກິລິຍາການຖອດຂໍ້ຄວາມແບບປີ້ນກັບ SuperScriptⅡ, ສາມາດເພີ່ມ 10% glycerol ແລະ incubated ຢູ່ທີ່ 45 ° C.ຖ້າ 1/10 ຂອງຜະລິດຕະພັນປະຕິກິລິຢາ reverse transcription ຖືກເພີ່ມໃສ່ PCR, ຫຼັງຈາກນັ້ນຄວາມເຂັ້ມຂົ້ນຂອງ glycerol ໃນປະຕິກິລິຍາຂະຫຍາຍແມ່ນ 0.4%, ເຊິ່ງບໍ່ພຽງພໍທີ່ຈະຍັບຍັ້ງ PCR.
5. ການປິ່ນປົວ RNaseH:
ການປິ່ນປົວປະຕິກິລິຍາສັງເຄາະ cDNA ກັບ RNaseH ກ່ອນ PCR ສາມາດເພີ່ມຄວາມອ່ອນໄຫວໄດ້.ສໍາລັບບາງແມ່ແບບ, ມັນຄິດວ່າ RNA ໃນປະຕິກິລິຍາສັງເຄາະ cDNA ປ້ອງກັນການຜູກມັດຂອງຜະລິດຕະພັນຂະຫຍາຍ, ໃນກໍລະນີນີ້ການປິ່ນປົວ RNaseH ສາມາດເພີ່ມຄວາມອ່ອນໄຫວ.ໂດຍທົ່ວໄປແລ້ວ, ການປິ່ນປົວ RNaseH ແມ່ນມີຄວາມຈໍາເປັນໃນເວລາທີ່ຂະຫຍາຍແມ່ແບບເປົ້າຫມາຍ cDNA ທີ່ມີຄວາມຍາວເຕັມທີ່ຍາວກວ່າ, ເຊັ່ນ: ໂຣກ tuberous scherosis II.ສໍາລັບແມ່ແບບທີ່ມີຄວາມຫຍຸ້ງຍາກນີ້, ການປິ່ນປົວ RNaseH ປັບປຸງສັນຍານທີ່ຜະລິດໂດຍ SuperScript II ຫຼື AMV-synthesized cDNA.ສໍາລັບປະຕິກິລິຍາ RT-PCR ສ່ວນໃຫຍ່, ການປິ່ນປົວ RNaseH ແມ່ນທາງເລືອກ, ເພາະວ່າຂັ້ນຕອນ PCR denaturation ຢູ່ທີ່ 95 ° C ໂດຍທົ່ວໄປແລ້ວ hydrolyzes RNA ໃນ RNA: DNA complex.
6. ການປັບປຸງວິທີການກວດຫາ RNA ຂະໜາດນ້ອຍ:
RT-PCR ມີຄວາມທ້າທາຍໂດຍສະເພາະເມື່ອມີ RNA ຈໍານວນນ້ອຍໆເທົ່ານັ້ນ.Glycogen ເພີ່ມເປັນຜູ້ໃຫ້ບໍລິການໃນລະຫວ່າງການແຍກ RNA ຊ່ວຍເພີ່ມຜົນຜະລິດຂອງຕົວຢ່າງຂະຫນາດນ້ອຍ.ສາມາດເພີ່ມ glycogen ທີ່ບໍ່ມີ RNase ໃນເວລາດຽວກັນກັບການເພີ່ມ Trizol.Glycogen ແມ່ນລະລາຍໃນນ້ໍາແລະສາມາດເກັບຮັກສາໄວ້ໃນໄລຍະນ້ໍາທີ່ມີ RNA ເພື່ອຊ່ວຍໃຫ້ມີຝົນຕົກຕໍ່ມາ.ສໍາລັບຕົວຢ່າງຫນ້ອຍກ່ວາ 50 ມລກຂອງເນື້ອເຍື່ອຫຼື 106 ຈຸລັງປູກຝັງ, ຄວາມເຂັ້ມຂຸ້ນທີ່ແນະນໍາຂອງ glycogen ທີ່ບໍ່ມີ RNase ແມ່ນ 250 μg / ml.
ການເພີ່ມ BSA acetylated ກັບປະຕິກິລິຢາ reverse transcription ໂດຍໃຊ້ SuperScript II ສາມາດເພີ່ມຄວາມອ່ອນໄຫວໄດ້, ແລະສໍາລັບ RNA ຂະຫນາດນ້ອຍ, ການຫຼຸດຜ່ອນປະລິມານຂອງ SuperScript II ແລະເພີ່ມ 40 ຫນ່ວຍຂອງ RNaseOut nuclease inhibitor ສາມາດເພີ່ມລະດັບການກວດພົບ.ຖ້າ glycogen ຖືກນໍາໃຊ້ໃນຂະບວນການແຍກ RNA, ມັນຍັງແນະນໍາໃຫ້ເພີ່ມ BSA ຫຼື RNase inhibitor ໃນເວລາໃຊ້ SuperScript II ສໍາລັບປະຕິກິລິຍາການຖອດຂໍ້ຄວາມຄືນ.
二、ເພີ່ມຄວາມສະເພາະຂອງ RT-PCR
1. ການສັງເຄາະ CND:
ການສັງເຄາະ cDNA ສາຍທໍາອິດສາມາດລິເລີ່ມໄດ້ໂດຍໃຊ້ສາມວິທີທີ່ແຕກຕ່າງກັນ, ຄວາມສະເພາະຂອງພີ່ນ້ອງທີ່ມີຜົນກະທົບຕໍ່ຈໍານວນແລະປະເພດຂອງ cDNA ທີ່ສັງເຄາະ.
ວິທີການ primer ແບບສຸ່ມແມ່ນສະເພາະຢ່າງຫນ້ອຍໃນສາມວິທີການ.Primers anneal ຢູ່ຫຼາຍສະຖານທີ່ຕະຫຼອດການຖອດຂໍ້ຄວາມ, ສ້າງ cDNAs ສັ້ນ, ຄວາມຍາວບາງສ່ວນ.ວິທີການນີ້ຖືກນໍາໃຊ້ເລື້ອຍໆເພື່ອໃຫ້ໄດ້ 5′ ລໍາດັບທ້າຍແລະໄດ້ຮັບ cDNA ຈາກແມ່ແບບ RNA ກັບພາກພື້ນຂອງໂຄງສ້າງຮອງຫຼືກັບສະຖານທີ່ສິ້ນສຸດທີ່ບໍ່ສາມາດ replicated ໂດຍ reverse transcriptase.ເພື່ອໃຫ້ໄດ້ cDNA ທີ່ຍາວທີ່ສຸດ, ອັດຕາສ່ວນຂອງ primers ກັບ RNA ໃນແຕ່ລະຕົວຢ່າງ RNA ຈໍາເປັນຕ້ອງໄດ້ຖືກກໍານົດໂດຍທາງກົງ.ຄວາມເຂັ້ມຂຸ້ນເລີ່ມຕົ້ນຂອງ primers ແບບສຸ່ມແມ່ນຕັ້ງແຕ່ 50 ຫາ 250 ng ຕໍ່ປະຕິກິລິຍາ 20 μl.ນັບຕັ້ງແຕ່ cDNA ສັງເຄາະຈາກ RNA ທັງຫມົດໂດຍໃຊ້ primers ແບບສຸ່ມແມ່ນ RNA ribosomal ຕົ້ນຕໍ, poly(A) + RNA ໂດຍທົ່ວໄປແມ່ນຖືກເລືອກເປັນແມ່ແບບ.
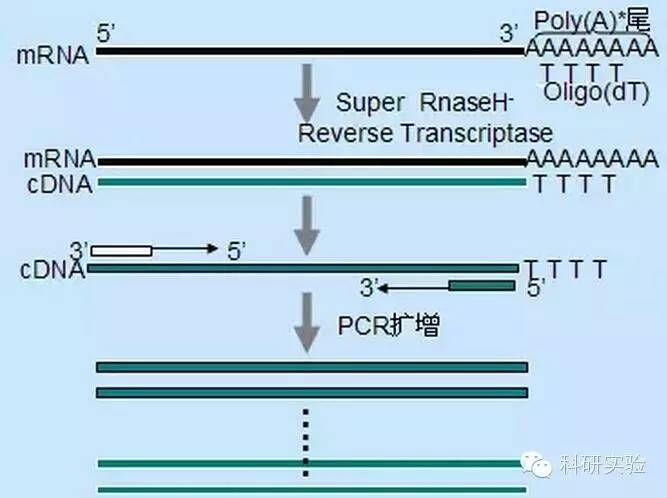
primers Oligo(dT) ແມ່ນສະເພາະຫຼາຍກ່ວາ primers ແບບສຸ່ມ.ມັນປະສົມກັບຫາງ poly(A) ທີ່ພົບເຫັນຢູ່ປາຍ 3′ ຂອງ mRNA eukaryotic ສ່ວນໃຫຍ່.ເນື່ອງຈາກວ່າ poly(A)+ RNA ແມ່ນປະມານ 1% ຫາ 2% ຂອງ RNA ທັງໝົດ, ປະລິມານ ແລະ ຄວາມຊັບຊ້ອນຂອງ cDNA ແມ່ນໜ້ອຍກວ່າກັບ primers ແບບສຸ່ມ.ເນື່ອງຈາກຄວາມສະເພາະສູງຂອງມັນ, oligo(dT) ໂດຍທົ່ວໄປແລ້ວບໍ່ຮຽກຮ້ອງໃຫ້ມີການເພີ່ມປະສິດທິພາບຂອງອັດຕາສ່ວນຂອງ RNA ກັບ primers ແລະການຄັດເລືອກ poly(A)+.ແນະນໍາໃຫ້ໃຊ້ 0.5μg oligo(dT) ຕໍ່ລະບົບຕິກິຣິຍາ20μl.oligo(dT)12-18 ແມ່ນເຫມາະສົມສໍາລັບ RT-PCR ຫຼາຍທີ່ສຸດ.ThermoScript RT-PCR System ສະຫນອງ oligo(dT)20 ເນື່ອງຈາກຄວາມຫມັ້ນຄົງຂອງຄວາມຮ້ອນທີ່ດີກວ່າສໍາລັບອຸນຫະພູມ incubation ສູງຂຶ້ນ.
Gene specific primers (GSP) ແມ່ນ primers ສະເພາະທີ່ສຸດສໍາລັບຂັ້ນຕອນ transcription reverse.GSP ແມ່ນສານຕ້ານອະນຸມູນອິດສະລະ oligonucleotide ທີ່ສາມາດປະສົມພັນກັບລໍາດັບເປົ້າຫມາຍ RNA ໂດຍສະເພາະ, ບໍ່ເຫມືອນກັບ primers random ຫຼື oligo (dT), ເຊິ່ງເຮັດໃຫ້ RNAs ທັງຫມົດ.ກົດລະບຽບດຽວກັນທີ່ໃຊ້ໃນການອອກແບບ primers PCR ໃຊ້ກັບການອອກແບບຂອງ GSP ໃນປະຕິກິລິຍາການຖອດຂໍ້ຄວາມແບບປີ້ນກັບກັນ.GSP ສາມາດເປັນລໍາດັບດຽວກັນກັບ primer amplification ທີ່ anneals ກັບ 3′-ທີ່ສຸດຂອງ mRNA, ຫຼື GSP ສາມາດຖືກອອກແບບເພື່ອ anneal downstream ຂອງ primer amplification reverse.ສໍາລັບບາງວິຊາທີ່ຂະຫຍາຍໃຫຍ່ຂື້ນ, ຫຼາຍກວ່າຫນຶ່ງ antisense primer ຕ້ອງໄດ້ຮັບການອອກແບບສໍາລັບ RT-PCR ສົບຜົນສໍາເລັດເນື່ອງຈາກວ່າໂຄງສ້າງຂັ້ນສອງຂອງ RNA ເປົ້າຫມາຍອາດຈະປ້ອງກັນບໍ່ໃຫ້ primer binding.ແນະນໍາໃຫ້ໃຊ້ 1 pmol antisense GSP ໃນປະຕິກິລິຍາສັງເຄາະສາຍທໍາອິດ 20 μl.
2. ເພີ່ມອຸນຫະພູມ incubation ສໍາລັບ reverse transcription:
ເພື່ອໃຊ້ປະໂຍດອັນເຕັມທີ່ຂອງຄວາມສະເພາະຂອງ GSP, ຄວນໃຊ້ transcriptase reverse ທີ່ມີ thermostability ສູງກວ່າ.Thermostable reverse transcriptases ສາມາດ incubated ໃນອຸນຫະພູມທີ່ສູງຂຶ້ນເພື່ອເພີ່ມຄວາມເຂັ້ມງວດຕິກິຣິຍາ.ຕົວຢ່າງ, ຖ້າ GSP ຊ້ອນທ້າຍຢູ່ທີ່ 55 ° C, ສະເພາະຂອງ GSP ຈະບໍ່ຖືກໃຊ້ຢ່າງເຕັມທີ່ຖ້າ AMV ຫຼື M-MLV ຖືກໃຊ້ສໍາລັບການຖອດຂໍ້ຄວາມຍ້ອນກັບທີ່ຄວາມເຂັ້ມງວດຕ່ໍາຂອງ 37 ° C.ຢ່າງໃດກໍ່ຕາມ, SuperScript II ແລະ ThermoScript ສາມາດຖືກປະຕິກິລິຍາຢູ່ທີ່ 50 ° C ຫຼືສູງກວ່າ, ເຊິ່ງຈະກໍາຈັດຜະລິດຕະພັນທີ່ບໍ່ສະເພາະທີ່ສ້າງຂຶ້ນໃນອຸນຫະພູມຕ່ໍາ.ສໍາລັບຄວາມສະເພາະສູງສຸດ, ການປະສົມ RNA / primer ສາມາດຖືກໂອນໂດຍກົງຈາກອຸນຫະພູມ denaturation 65 ° C ໄປສູ່ອຸນຫະພູມ incubation transcription reverse ແລະເພີ່ມເຂົ້າໄປໃນການປະສົມຕິກິຣິຍາ 2 × pre- warmed (ການເລີ່ມຕົ້ນການສັງເຄາະ cDNA ຮ້ອນ).ນີ້ຊ່ວຍປ້ອງກັນການຈັບຄູ່ຖານ intermolecular ໃນອຸນຫະພູມຕ່ໍາ.ການຫັນປ່ຽນອຸນຫະພູມຫຼາຍອັນທີ່ຕ້ອງການສໍາລັບ RT-PCR ສາມາດງ່າຍດາຍໄດ້ໂດຍການໃຊ້ເຄື່ອງວົງຈອນຄວາມຮ້ອນ.
3. ຫຼຸດຜ່ອນການປົນເປື້ອນ DNA ພັນທຸ ກຳ:
ຄວາມຫຍຸ້ງຍາກທີ່ອາດຈະພົບກັບ RT-PCR ແມ່ນການປົນເປື້ອນຂອງ DNA ພັນທຸ ກຳ ໃນ RNA.ການນໍາໃຊ້ວິທີການແຍກ RNA ທີ່ດີ, ເຊັ່ນ Trizol Reagent, ຈະຫຼຸດຜ່ອນຈໍານວນຂອງ DNA genomic ທີ່ປົນເປື້ອນການກະກຽມ RNA.ເພື່ອຫຼີກເວັ້ນການຜະລິດຕະພັນທີ່ມາຈາກ DNA ພັນທຸ ກຳ, RNA ສາມາດຖືກປະຕິບັດດ້ວຍການຂະຫຍາຍລະດັບ DNase I ເພື່ອເອົາ DNA ທີ່ປົນເປື້ອນອອກກ່ອນການຖອດລະຫັດຄືນ.ການຍ່ອຍອາຫານຂອງ DNase I ໄດ້ຖືກຢຸດໂດຍການອົບຕົວຢ່າງໃນ 2.0 mM EDTA ເປັນເວລາ 10 ນາທີທີ່ອຸນຫະພູມ 65 ອົງສາ.EDTA ສາມາດ chelate magnesium ions, ປ້ອງກັນບໍ່ໃຫ້ magnesium ion-dependent RNA hydrolysis ໃນອຸນຫະພູມສູງ.
ເພື່ອແຍກ cDNA ຂະຫຍາຍອອກຈາກການປົນເປື້ອນຜະລິດຕະພັນຂະຫຍາຍພັນ DNA, primers ສາມາດຖືກອອກແບບທີ່ແຕ່ລະ anneal ເພື່ອແຍກ exons.ຜະລິດຕະພັນ PCR ທີ່ມາຈາກ cDNA ຈະສັ້ນກວ່າຜະລິດຕະພັນທີ່ມາຈາກ DNA ພັນທຸ ກຳ ທີ່ປົນເປື້ອນ.ນອກຈາກນັ້ນ, ການທົດລອງການຄວບຄຸມໂດຍບໍ່ມີການຖອດຖອນຄືນໄດ້ຖືກປະຕິບັດຢູ່ໃນແຕ່ລະແມ່ແບບ RNA ເພື່ອກໍານົດວ່າຊິ້ນສ່ວນທີ່ໄດ້ຮັບມາຈາກ DNA ຫຼື cDNA genomic.ຜະລິດຕະພັນ PCR ທີ່ໄດ້ຮັບໂດຍບໍ່ມີການຖອດຖອນຄືນແມ່ນໄດ້ມາຈາກ genome.
ເວລາປະກາດ: 16-05-2023








