ໃນການທົດລອງ qPCR, ການອອກແບບ primer ຍັງເປັນການເຊື່ອມໂຍງທີ່ສໍາຄັນ.ບໍ່ວ່າຈະເປັນ primers ທີ່ເຫມາະສົມຫຼືບໍ່ແມ່ນກ່ຽວຂ້ອງຢ່າງໃກ້ຊິດກັບວ່າປະສິດທິພາບການຂະຫຍາຍໄດ້ບັນລຸມາດຕະຖານ, ບໍ່ວ່າຈະເປັນຜະລິດຕະພັນຂະຫຍາຍໃຫຍ່ຂື້ນແມ່ນສະເພາະ, ແລະຜົນໄດ້ຮັບການທົດລອງ.
ດັ່ງນັ້ນວິທີການເຮັດໃຫ້ຄວາມສະເພາະຂອງ primer qPCR ດີກວ່າ?ປະສິດທິພາບການຂະຫຍາຍສູງ?
ມື້ນີ້, ພວກເຮົາຈະພາທ່ານໄປອອກແບບ primer qPCR ຮ່ວມກັນ, ແລະໃຫ້ການອອກແບບ primer qPCR ກາຍເປັນທັກສະທີ່ມີປະສິດທິພາບໃນການທົດລອງ.
ໃນເວລາທີ່ການອອກແບບ primers qPCR, ໂດຍປົກກະຕິຈະເອົາໃຈໃສ່ກັບຈຸດຕໍ່ໄປນີ້: primers ຄວນຖືກອອກແບບໃນທົ່ວ introns ເທົ່າທີ່ເປັນໄປໄດ້, ຄວາມຍາວຂອງຜະລິດຕະພັນຄວນຈະເປັນ 100-300 bp, ຄ່າ Tm ຄວນໃກ້ຊິດທີ່ສຸດເທົ່າທີ່ເປັນໄປໄດ້ 60 ° C, ແລະ primers ເທິງນ້ໍາແລະລຸ່ມຄວນຈະໃກ້ຊິດທີ່ສຸດເທົ່າທີ່ເປັນໄປໄດ້, ແລະການສິ້ນສຸດຂອງ primer ຄວນຈະເປັນ G ຫຼື C, ແລະອື່ນໆ.
1. ການອອກແບບ primers spanning intros
ໃນເວລາທີ່ການອອກແບບ primers qPCR, ການເລືອກ primers ທີ່ອອກແບບໃນທົ່ວ introns ສາມາດປ້ອງກັນບໍ່ໃຫ້ແມ່ແບບ gDNA ຖືກຂະຫຍາຍ, ແລະຜະລິດຕະພັນທັງຫມົດແມ່ນມາຈາກການຂະຫຍາຍຂອງ cDNA, ດັ່ງນັ້ນການກໍາຈັດອິດທິພົນຂອງການປົນເປື້ອນ gDNA.
2. ຄວາມຍາວຂອງ primer
ຄວາມຍາວ primer ໂດຍທົ່ວໄປແມ່ນຢູ່ລະຫວ່າງ 18-30 nt, ແລະຄວາມຍາວຂອງຜະລິດຕະພັນ amplification ຄວນໄດ້ຮັບການຄວບຄຸມລະຫວ່າງ 100-300 bp ເທົ່າທີ່ເປັນໄປໄດ້.
ຖ້າ primer ສັ້ນເກີນໄປ, ມັນຈະນໍາໄປສູ່ການຂະຫຍາຍທີ່ບໍ່ສະເພາະ, ແລະຖ້າມັນຍາວເກີນໄປ, ມັນຈະສ້າງໂຄງສ້າງຮອງໄດ້ຢ່າງງ່າຍດາຍ (ເຊັ່ນໂຄງສ້າງ hairpin).ຖ້າຜະລິດຕະພັນຂະຫຍາຍໃຫຍ່ຂື້ນຍາວເກີນໄປ, ມັນບໍ່ເຫມາະສົມສໍາລັບປະຕິກິລິຍາຂອງໂພລີເມີເຣດ, ເຊິ່ງຈະສົ່ງຜົນກະທົບຕໍ່ປະສິດທິພາບຂອງ PCR amplification.
3. ເນື້ອຫາ GC ແລະມູນຄ່າ Tm
ເນື້ອໃນ GC ຂອງ primers ຄວນໄດ້ຮັບການຄວບຄຸມລະຫວ່າງ 40% ແລະ 60%.ຖ້າມັນສູງເກີນໄປຫຼືຕ່ໍາເກີນໄປ, ມັນບໍ່ເຫມາະສົມສໍາລັບການລິເລີ່ມຕິກິຣິຍາ.ເນື້ອໃນ GC ຂອງ primers ໄປຂ້າງຫນ້າແລະປີ້ນກັບກັນຄວນຈະຢູ່ໃກ້ກັນເພື່ອໃຫ້ໄດ້ຄ່າ Tm ດຽວກັນແລະອຸນຫະພູມ annealing.
ຄ່າ Tm ຄວນຢູ່ລະຫວ່າງ 55-65 °C ເທົ່າທີ່ເປັນໄປໄດ້, ໂດຍທົ່ວໄປແລ້ວແມ່ນປະມານ 60 ° C, ແລະຄ່າ Tm ຂອງຕົ້ນນ້ໍາແລະລຸ່ມຄວນຈະໃກ້ຊິດເທົ່າທີ່ເປັນໄປໄດ້, ດີກວ່າບໍ່ເກີນ 4 ° C.
4. ຫຼີກເວັ້ນການເລືອກ A ຢູ່ປາຍ 3′ ຂອງ primer
ເມື່ອ 3′ ປາຍຂອງ primer ບໍ່ກົງກັນ, ມີຄວາມແຕກຕ່າງຢ່າງຫຼວງຫຼາຍໃນປະສິດທິພາບການສັງເຄາະຂອງຖານທີ່ແຕກຕ່າງກັນ.ເມື່ອຖານສຸດທ້າຍແມ່ນ A, ມັນຍັງສາມາດລິເລີ່ມການສັງເຄາະລະບົບຕ່ອງໂສ້ເຖິງແມ່ນວ່າໃນກໍລະນີຂອງການບໍ່ກົງກັນ, ແລະໃນເວລາທີ່ພື້ນຖານສຸດທ້າຍແມ່ນ T ເມື່ອ, ປະສິດທິພາບຂອງການ induction ບໍ່ກົງກັນແມ່ນຫຼຸດລົງຢ່າງຫຼວງຫຼາຍ.ສະນັ້ນ, ພະຍາຍາມຫຼີກລ່ຽງການເລືອກ A ຢູ່ປາຍ 3′ ຂອງ primer, ແລະ ຄວນເລືອກ T.
ຖ້າມັນເປັນ primer probe, 5′ ປາຍຂອງ probe ບໍ່ສາມາດເປັນ G, ເພາະວ່າເຖິງແມ່ນວ່າໃນເວລາທີ່ຖານ G ດຽວຈະເຊື່ອມຕໍ່ກັບກຸ່ມນັກຂ່າວ FAM fluorescent, G ຍັງສາມາດດັບສັນຍານ fluorescent ທີ່ປ່ອຍອອກມາໂດຍກຸ່ມ FAM, ສົ່ງຜົນໃຫ້ຜົນໄດ້ຮັບທາງລົບທີ່ບໍ່ຖືກຕ້ອງ.ປາກົດ.
5. ການແຜ່ກະຈາຍພື້ນຖານ
ການແຜ່ກະຈາຍຂອງສີ່ຖານໃນ primer ແມ່ນດີກວ່າແບບສຸ່ມ, ຫຼີກເວັ້ນຫຼາຍກວ່າ 3 G ຫຼື C ຕິດຕໍ່ກັນໃນຕອນທ້າຍຂອງ 3′, ແລະຫຼາຍກວ່າ 3 ຕິດຕໍ່ກັນ.G ຫຼື C ແມ່ນງ່າຍທີ່ຈະສ້າງການຈັບຄູ່ໃນພາກພື້ນລໍາດັບ GC ທີ່ອຸດົມສົມບູນ.
6. ພາກພື້ນການອອກແບບ primer ຄວນຫຼີກເວັ້ນໂຄງສ້າງຮອງທີ່ສັບສົນ.
ໂຄງສ້າງຂັ້ນສອງທີ່ສ້າງຂຶ້ນໂດຍສາຍດຽວຂອງຜະລິດຕະພັນຂະຫຍາຍໃຫຍ່ຂື້ນຈະສົ່ງຜົນກະທົບຕໍ່ຄວາມກ້າວຫນ້າຂອງ PCR.ໂດຍການຄາດເດົາວ່າມີໂຄງສ້າງຂັ້ນສອງໃນລໍາດັບເປົ້າຫມາຍລ່ວງຫນ້າ, ພະຍາຍາມຫຼີກເວັ້ນພາກພື້ນນີ້ໃນການອອກແບບ primers.
7. primers ຕົວເອງແລະລະຫວ່າງ primers ຄວນພະຍາຍາມຫຼີກລ້ຽງການເສີມສ້າງພື້ນຖານຕິດຕໍ່ກັນ.
ບໍ່ສາມາດມີພື້ນຖານ 4 ຕິດຕໍ່ກັນລະຫວ່າງ primer ຕົວມັນເອງແລະ primer.primer ຕົວຂອງມັນເອງບໍ່ຄວນມີລໍາດັບເສີມ, ຖ້າບໍ່ດັ່ງນັ້ນມັນຈະພັບຕົວມັນເອງເພື່ອສ້າງໂຄງສ້າງ hairpin, ເຊິ່ງຈະສົ່ງຜົນກະທົບຕໍ່ການປະສົມປະສານຂອງ primer ແລະ template.
ລໍາດັບເສີມບໍ່ສາມາດມີຢູ່ລະຫວ່າງ primers ເທິງນ້ໍາແລະລຸ່ມນ້ໍາ.ການປະສົມປະສານລະຫວ່າງ primers ຈະຜະລິດ primer dimers, ເຊິ່ງຈະຫຼຸດລົງປະສິດທິພາບ PCR ແລະແມ້ກະທັ້ງຜົນກະທົບຕໍ່ຄວາມຖືກຕ້ອງຂອງປະລິມານ.ຖ້າໂຄງສ້າງ primer-dimer ແລະ hairpin ແມ່ນຫຼີກລ້ຽງບໍ່ໄດ້, ຄ່າ △G ບໍ່ຄວນສູງເກີນໄປ (ຄວນຈະຫນ້ອຍກວ່າ 4.5 kcal / mol).
8. primers ຂະຫຍາຍຜະລິດຕະພັນສະເພາະເປົ້າຫມາຍ.
ເປົ້າຫມາຍສຸດທ້າຍຂອງການກວດສອບ qPCR ແມ່ນເພື່ອເຂົ້າໃຈຄວາມອຸດົມສົມບູນຂອງ gene ເປົ້າຫມາຍ.ຖ້າການຂະຫຍາຍທີ່ບໍ່ສະເພາະເກີດຂຶ້ນ, ປະລິມານຈະບໍ່ຖືກຕ້ອງ.ດັ່ງນັ້ນ, ຫຼັງຈາກ primers ຖືກອອກແບບມາ, ພວກເຂົາຕ້ອງໄດ້ຮັບການທົດສອບໂດຍ BLAST, ແລະຄວາມສະເພາະຂອງຜະລິດຕະພັນໄດ້ຖືກປຽບທຽບໃນຖານຂໍ້ມູນລໍາດັບ.
ຕໍ່ໄປ, ພວກເຮົາເອົາ gene GAS6 (Growth arrest specific 6) ຂອງມະນຸດເປັນຕົວຢ່າງເພື່ອອອກແບບ primers qPCR.
01 gene query
Homo GAS6ຜ່ານ NCBI.ໃນທີ່ນີ້, ພວກເຮົາຄວນເອົາໃຈໃສ່ກັບການປຽບທຽບຊື່ພັນທຸກໍາແລະຊະນິດພັນເພື່ອຮັບປະກັນວ່າພວກມັນສອດຄ່ອງ.
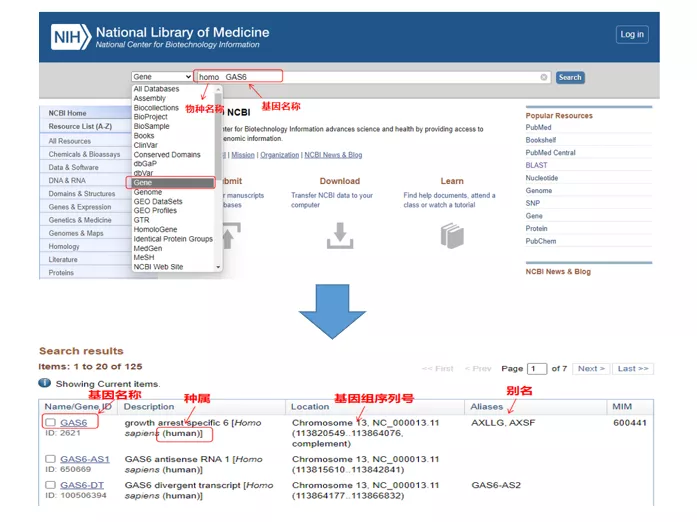 02 ຊອກຫາລໍາດັບ gene
02 ຊອກຫາລໍາດັບ gene
(1) ຖ້າລໍາດັບເປົ້າຫມາຍແມ່ນ DNA genomic, ເລືອກອັນທໍາອິດ, ເຊິ່ງເປັນລໍາດັບ DNA genomic ຂອງ gene.
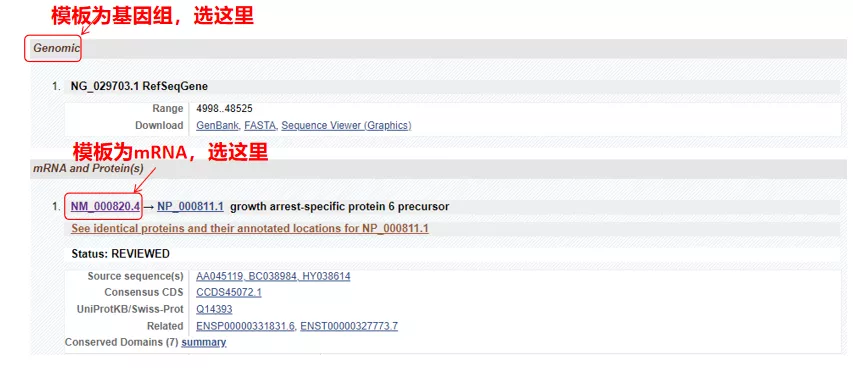 (2) ຖ້າລໍາດັບເປົ້າຫມາຍແມ່ນ mRNA, ເລືອກອັນທີສອງ.ຫຼັງຈາກທີ່ເຂົ້າມາ, ໃຫ້ຄລິກໃສ່ "CDS" ໃນຕາຕະລາງຂ້າງລຸ່ມນີ້.ລໍາດັບພື້ນຖານສີນ້ໍາຕານແມ່ນລໍາດັບລະຫັດຂອງ gene.
(2) ຖ້າລໍາດັບເປົ້າຫມາຍແມ່ນ mRNA, ເລືອກອັນທີສອງ.ຫຼັງຈາກທີ່ເຂົ້າມາ, ໃຫ້ຄລິກໃສ່ "CDS" ໃນຕາຕະລາງຂ້າງລຸ່ມນີ້.ລໍາດັບພື້ນຖານສີນ້ໍາຕານແມ່ນລໍາດັບລະຫັດຂອງ gene.
 03 ການອອກແບບ primers
03 ການອອກແບບ primers
ໃສ່ສ່ວນຕິດຕໍ່ Primer-BLAST
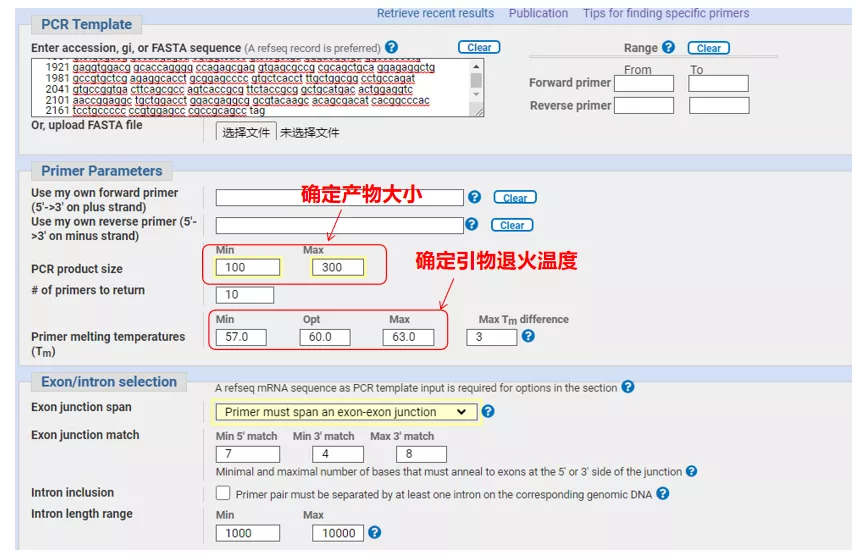
 ໃສ່ເລກລໍາດັບ gene ຫຼືລໍາດັບໃນຮູບແບບ Fasta ຢູ່ເບື້ອງຊ້າຍເທິງ, ແລະຕື່ມຂໍ້ມູນໃສ່ໃນຕົວກໍານົດການທີ່ກ່ຽວຂ້ອງ.
ໃສ່ເລກລໍາດັບ gene ຫຼືລໍາດັບໃນຮູບແບບ Fasta ຢູ່ເບື້ອງຊ້າຍເທິງ, ແລະຕື່ມຂໍ້ມູນໃສ່ໃນຕົວກໍານົດການທີ່ກ່ຽວຂ້ອງ.
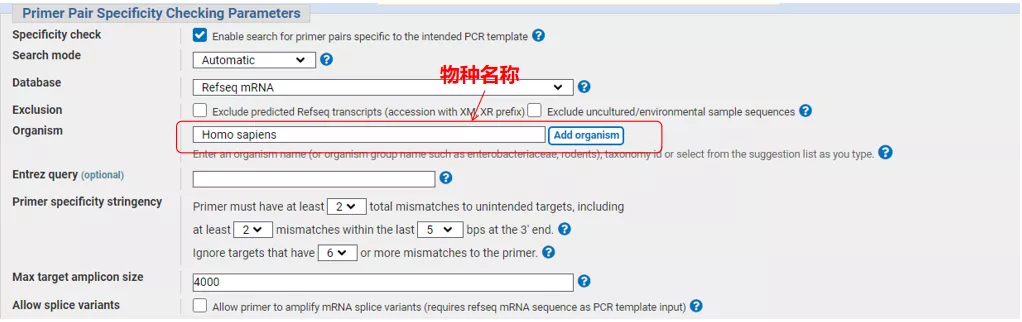
ໃຫ້ຄລິກໃສ່ "Get primers" ແລະ NCBI ຈະປາກົດຂຶ້ນເພື່ອບອກທ່ານວ່າການເລືອກພາລາມິເຕີດັ່ງກ່າວຈະຖືກຂະຫຍາຍໄປສູ່ຕົວແປອື່ນໆ.ພວກເຮົາສາມາດກວດສອບການ splicing variants ທີ່ແຕກຕ່າງກັນແລະສົ່ງໃຫ້ເຂົາເຈົ້າໄດ້ຮັບຄູ່ primer ທີ່ເຫມາະສົມ (ດັ່ງທີ່ສະແດງໃຫ້ເຫັນຢູ່ໃນຮູບຂ້າງລຸ່ມນີ້).ຂະບວນການນີ້ອາດຈະໃຊ້ເວລາຫຼາຍສິບວິນາທີເພື່ອດໍາເນີນການ.
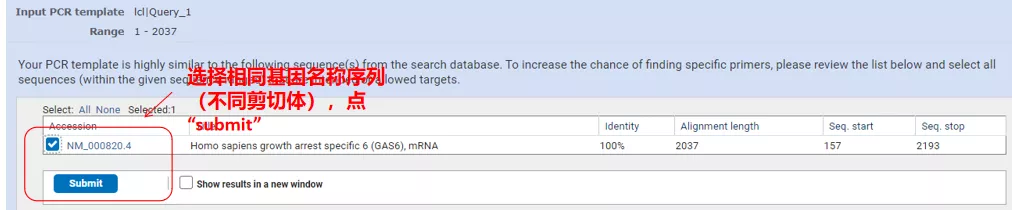
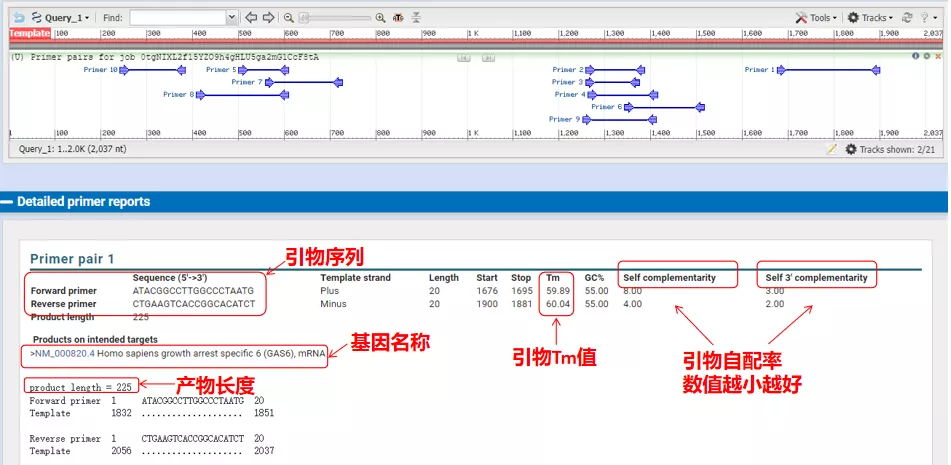 ອຸນຫະພູມຂອງຄູ່ primer ເຫຼົ່ານີ້ແມ່ນທັງຫມົດປະມານ 60°C.ອີງຕາມຈຸດປະສົງຂອງການທົດລອງ, ເລືອກເອົາ primers ທີ່ມີຄວາມຍາວປານກາງ, ສະເພາະທີ່ດີແລະຫນ້ອຍການເສີມຕົນເອງຂອງ primers ສໍາລັບການທົດລອງ, ແລະອັດຕາຜົນສໍາເລັດແມ່ນຂ້ອນຂ້າງສູງ!
ອຸນຫະພູມຂອງຄູ່ primer ເຫຼົ່ານີ້ແມ່ນທັງຫມົດປະມານ 60°C.ອີງຕາມຈຸດປະສົງຂອງການທົດລອງ, ເລືອກເອົາ primers ທີ່ມີຄວາມຍາວປານກາງ, ສະເພາະທີ່ດີແລະຫນ້ອຍການເສີມຕົນເອງຂອງ primers ສໍາລັບການທົດລອງ, ແລະອັດຕາຜົນສໍາເລັດແມ່ນຂ້ອນຂ້າງສູງ!
04 ການກວດສອບສະເພາະ Primer
ໃນຄວາມເປັນຈິງ, ນອກເຫນືອຈາກການອອກແບບ primers, Primer-Blast ຍັງສາມາດປະເມີນ primers ທີ່ພວກເຮົາອອກແບບເອງ.ກັບຄືນໄປຫາຫນ້າການອອກແບບ primer, ເຂົ້າໄປໃນ primers ເທິງແລະລຸ່ມທີ່ພວກເຮົາອອກແບບ, ແລະຕົວກໍານົດການອື່ນໆຈະບໍ່ຖືກປັບ.ຫຼັງຈາກການສົ່ງ, ທ່ານສາມາດເບິ່ງວ່າຄູ່ຂອງ primers ຍັງມີຢູ່ໃນພັນທຸກໍາອື່ນໆ.ຖ້າພວກເຂົາທັງຫມົດຖືກສະແດງຢູ່ໃນ gene ທີ່ພວກເຮົາຕ້ອງການຂະຫຍາຍ, ສະແດງໃຫ້ເຫັນວ່າຄວາມສະເພາະຂອງ primers ຄູ່ນີ້ແມ່ນດີເລີດ!(ຕົວຢ່າງ, ນີ້ແມ່ນຜົນພຽງແຕ່ຂອງການສອບຖາມ primer!)
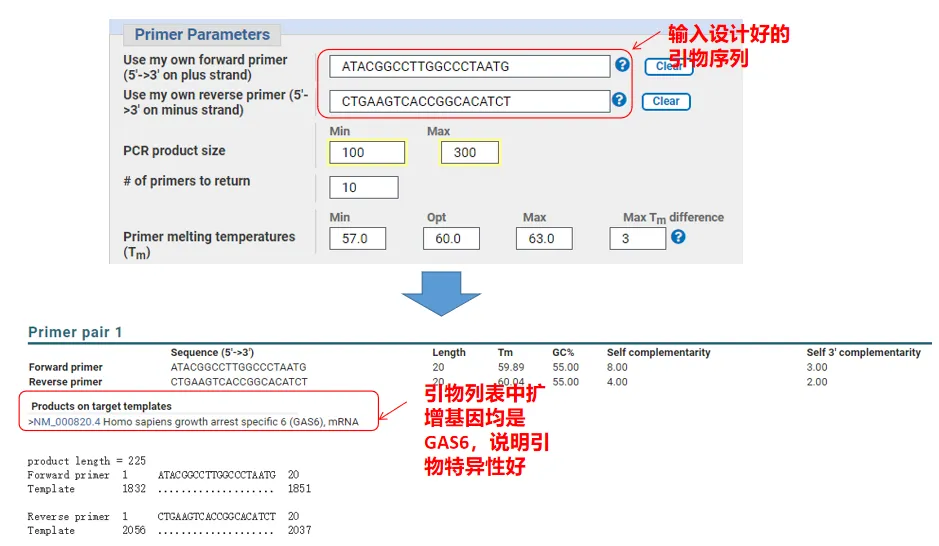
05 ການຕັດສິນຄຸນນະພາບ Primer
primer ປະເພດໃດແດ່ແມ່ນ primer "ທີ່ສົມບູນແບບ" ທີ່ປະສົມປະສານ "ປະສິດທິພາບການຂະຫຍາຍໄດ້ມາດຕະຖານ", "ຄຸນລັກສະນະຜະລິດຕະພັນຂະຫຍາຍ", ແລະ "ຜົນການທົດລອງທີ່ເຊື່ອຖືໄດ້"?
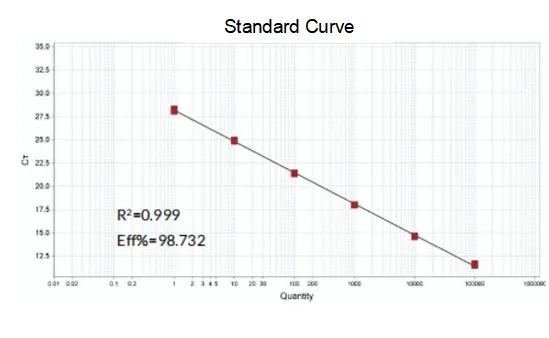 ປະສິດທິພາບການຂະຫຍາຍ
ປະສິດທິພາບການຂະຫຍາຍ
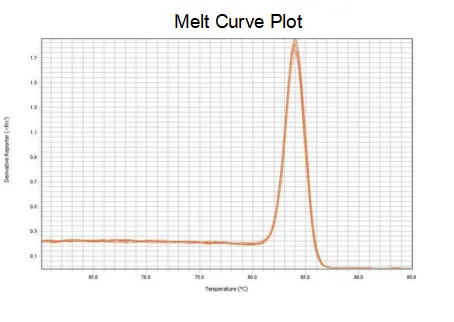 ເສັ້ນໂຄ້ງ melting
ເສັ້ນໂຄ້ງ melting
ປະສິດທິພາບການຂະຫຍາຍຂອງ primers ບັນລຸ 90%-110%, ຊຶ່ງຫມາຍຄວາມວ່າປະສິດທິພາບການຂະຫຍາຍແມ່ນດີ, ແລະເສັ້ນໂຄ້ງ melting ມີຈຸດສູງສຸດດຽວແລະປົກກະຕິແລ້ວ Tm> 80 ° C, ຊຶ່ງຫມາຍຄວາມວ່າສະເພາະການຂະຫຍາຍແມ່ນດີ.
ຜະລິດຕະພັນທີ່ກ່ຽວຂ້ອງ:
ເວລາຈິງ PCR Easy-SYBR GREEN I
ເວລາຈິງ PCR Easy-Taqman
ເວລາປະກາດ: Feb-10-2023








