ວັດສະດຸເລີ່ມຕົ້ນ: RNA
ການຖອດຂໍ້ຄວາມແບບປີ້ນກັບປະລິມານ PCR (RT-qPCR) ແມ່ນວິທີການທົດລອງທີ່ໃຊ້ໃນການທົດລອງ PCR ໂດຍໃຊ້ RNA ເປັນວັດສະດຸເລີ່ມຕົ້ນ.ໃນວິທີການນີ້, RNA ທັງຫມົດຫຼື messenger RNA (mRNA) ທໍາອິດຖືກຖ່າຍທອດເຂົ້າໄປໃນ DNA ເສີມ (cDNA) ດ້ວຍການຖອດຂໍ້ຄວາມແບບປີ້ນກັບກັນ.ຫຼັງຈາກນັ້ນ, ປະຕິກິລິຍາ qPCR ໄດ້ຖືກປະຕິບັດໂດຍໃຊ້ cDNA ເປັນແມ່ແບບ.RT-qPCR ໄດ້ຖືກນໍາໃຊ້ໃນຫຼາຍໆຄໍາຮ້ອງສະຫມັກຊີວະວິທະຍາໂມເລກຸນ, ລວມທັງການວິເຄາະການສະແດງອອກຂອງເຊື້ອ, ການກວດສອບການແຊກແຊງ RNA, ການກວດສອບ microarray, ການກວດຫາເຊື້ອພະຍາດ, ການທົດສອບພັນທຸກໍາ, ແລະການຄົ້ນຄວ້າພະຍາດ.
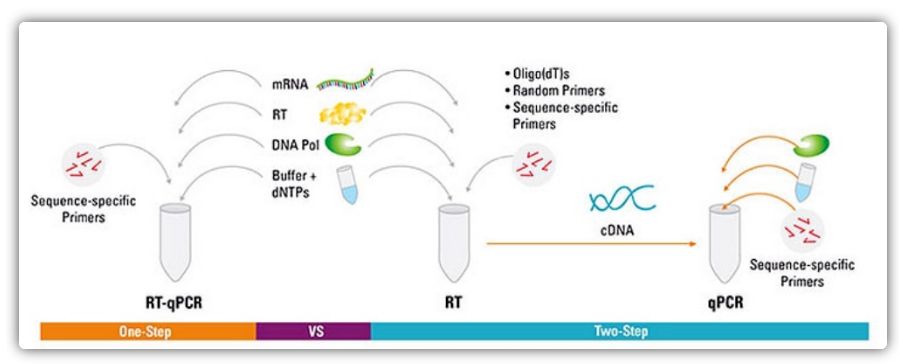
ວິທີການຫນຶ່ງຂັ້ນຕອນແລະສອງຂັ້ນຕອນສໍາລັບ RT-qPCR
RT-qPCR ສາມາດເຮັດໄດ້ໂດຍວິທີການຫນຶ່ງຂັ້ນຕອນຫຼືສອງຂັ້ນຕອນ.RT-qPCR ຂັ້ນຕອນດຽວປະສົມປະສານການຖອດຂໍ້ຄວາມແບບປີ້ນກັບແລະການຂະຫຍາຍ PCR, ອະນຸຍາດໃຫ້ reverse transcriptase ແລະ DNA polymerase ສໍາເລັດຕິກິຣິຍາໃນທໍ່ດຽວກັນພາຍໃຕ້ເງື່ອນໄຂ buffer ດຽວກັນ.RT-qPCR ຂັ້ນຕອນດຽວຕ້ອງການໃຊ້ primers ສະເພາະຕາມລໍາດັບ.ໃນສອງຂັ້ນຕອນ RT-qPCR, reverse transcription ແລະ PCR amplification ແມ່ນດໍາເນີນຢູ່ໃນສອງທໍ່, ການນໍາໃຊ້ buffers optimized ທີ່ແຕກຕ່າງກັນ, ເງື່ອນໄຂຕິກິຣິຍາ, ແລະຍຸດທະສາດການອອກແບບ primer.
| ຂໍ້ໄດ້ປຽບ | ຂໍ້ເສຍ | |
| ຂັ້ນຕອນຫນຶ່ງ | ວິທີການນີ້ມີຄວາມຜິດພາດໃນການທົດລອງຫນ້ອຍລົງຍ້ອນວ່າປະຕິກິລິຍາທັງສອງແມ່ນເຮັດຢູ່ໃນທໍ່ດຽວ
ຂັ້ນຕອນການທໍ່ທໍ່ໜ້ອຍລົງ ຫຼຸດຜ່ອນຄວາມສ່ຽງຕໍ່ການປົນເປື້ອນ
ເຫມາະສົມສໍາລັບການຂະຫຍາຍ / ການຄັດເລືອກໂດຍຜ່ານການສູງ, ໄວແລະການແຜ່ພັນ | ປະຕິກິລິຍາສອງຂັ້ນຕອນບໍ່ສາມາດຖືກປັບແຕ່ງແຍກຕ່າງຫາກ
ເນື່ອງຈາກເງື່ອນໄຂປະຕິກິລິຢາຖືກປະນີປະນອມໂດຍການລວມຕິກິຣິຍາສອງຂັ້ນຕອນ, ຄວາມອ່ອນໄຫວບໍ່ດີເທົ່າກັບວິທີການສອງຂັ້ນຕອນ.
ຈໍານວນເປົ້າຫມາຍທີ່ກວດພົບໂດຍຕົວຢ່າງດຽວແມ່ນຫນ້ອຍ |
| ສອງຂັ້ນຕອນ | ຄວາມສາມາດໃນການສ້າງຫ້ອງສະຫມຸດ cDNA ທີ່ຫມັ້ນຄົງທີ່ສາມາດເກັບຮັກສາໄວ້ເປັນເວລາດົນນານແລະນໍາໃຊ້ໃນຫຼາຍຕິກິລິຍາ
genes ເປົ້າຫມາຍແລະ genes ອ້າງອິງສາມາດຂະຫຍາຍໄດ້ຈາກຫ້ອງສະຫມຸດ cDNA ດຽວກັນໂດຍບໍ່ຈໍາເປັນຕ້ອງມີຫ້ອງສະຫມຸດ cDNA ຫຼາຍ.
ປະຕິກິລິຍາ buffers ແລະເງື່ອນໄຂຕິກິຣິຍາທີ່ເຮັດໃຫ້ການເພີ່ມປະສິດທິພາບຂອງປະຕິກິລິຍາດຽວ
ການຄັດເລືອກແບບຍືດຫຍຸ່ນຂອງເງື່ອນໄຂຜົນກະທົບຕໍ່ | ການນໍາໃຊ້ທໍ່ຫຼາຍ, ແລະຂັ້ນຕອນການທໍ່ຫຼາຍເພີ່ມຄວາມສ່ຽງຕໍ່ການປົນເປື້ອນ DNA, ແລະໃຊ້ເວລາຫຼາຍ.
ຮຽກຮ້ອງໃຫ້ມີການເພີ່ມປະສິດທິພາບຫຼາຍກວ່າວິທີການຫນຶ່ງຂັ້ນຕອນ |
ຜະລິດຕະພັນທີ່ກ່ຽວຂ້ອງ:
RT-qPCR Easyᵀᴹ (ຂັ້ນຕອນດຽວ)-SYBR Green I
RT-qPCR Easyᵀᴹ (ຂັ້ນຕອນຫນຶ່ງ)-Taqman
RT Easyᵀᴹ I Master Premix ສໍາລັບການສັງເຄາະ CDNA ທໍາອິດ
ເວລາຈິງ PCR Easyᵀᴹ-SYBR Green I Kit
ການຄັດເລືອກຂອງ RNA ແລະ mRNA ທັງຫມົດ
ເມື່ອອອກແບບການທົດລອງ RT-qPCR, ມັນເປັນສິ່ງສໍາຄັນທີ່ຈະຕັດສິນໃຈວ່າຈະໃຊ້ RNA ທັງຫມົດຫຼື mRNA ບໍລິສຸດເປັນແມ່ແບບສໍາລັບການຖອດຂໍ້ຄວາມແບບປີ້ນກັບກັນ.ເຖິງແມ່ນວ່າ mRNA ອາດຈະສາມາດສະຫນອງຄວາມອ່ອນໄຫວສູງກວ່າເລັກນ້ອຍ, RNA ທັງຫມົດຍັງຖືກນໍາໃຊ້ເລື້ອຍໆ.ເຫດຜົນສໍາລັບການນີ້ແມ່ນວ່າ RNA ທັງຫມົດມີປະໂຫຍດທີ່ສໍາຄັນກວ່າເປັນອຸປະກອນການເລີ່ມຕົ້ນຫຼາຍກ່ວາ mRNA.ຫນ້າທໍາອິດ, ຂະບວນການຮຽກຮ້ອງໃຫ້ມີຂັ້ນຕອນການຊໍາລະລ້າງຫນ້ອຍລົງ, ເຊິ່ງຮັບປະກັນການຟື້ນຕົວຂອງແມ່ແບບໃນປະລິມານທີ່ດີກວ່າແລະການເຮັດໃຫ້ຜົນໄດ້ຮັບປົກກະຕິດີກວ່າທີ່ຈະເລີ່ມຕົ້ນຈໍານວນເຊນ.ອັນທີສອງ, ມັນຫລີກລ້ຽງຂັ້ນຕອນການເສີມສ້າງ mRNA, ເຊິ່ງສາມາດຫລີກລ້ຽງຄວາມເປັນໄປໄດ້ຂອງຜົນໄດ້ຮັບທີ່ຜິດພາດຍ້ອນການຟື້ນຕົວຂອງ mRNA ທີ່ແຕກຕ່າງກັນ.ໂດຍລວມແລ້ວ, ເນື່ອງຈາກວ່າໃນຄໍາຮ້ອງສະຫມັກສ່ວນໃຫຍ່, ປະລິມານທີ່ກ່ຽວຂ້ອງຂອງ gene ເປົ້າຫມາຍແມ່ນມີຄວາມສໍາຄັນຫຼາຍກ່ວາຄວາມອ່ອນໄຫວຢ່າງແທ້ຈິງຂອງການກວດຫາ, RNA ທັງຫມົດແມ່ນເຫມາະສົມກວ່າໃນກໍລະນີຫຼາຍທີ່ສຸດ.
ການຖອດຂໍ້ຄວາມແບບປີ້ນກັບ primer
ໃນວິທີການສອງຂັ້ນຕອນ, ສາມວິທີທີ່ແຕກຕ່າງກັນສາມາດຖືກນໍາໃຊ້ເພື່ອ primers ປະຕິກິລິຍາ cDNA: oligo(dT) primers, primers random, ຫຼື sequence-specific primers.ໂດຍປົກກະຕິ, primers oligo(dT) ແລະ primers ແບບສຸ່ມແມ່ນໃຊ້ປະສົມປະສານ.primers ເຫຼົ່ານີ້ເຊື່ອມກັບແມ່ແບບ mRNA strand ແລະສະຫນອງ reverse transcriptase ກັບຈຸດເລີ່ມຕົ້ນສໍາລັບການສັງເຄາະ.
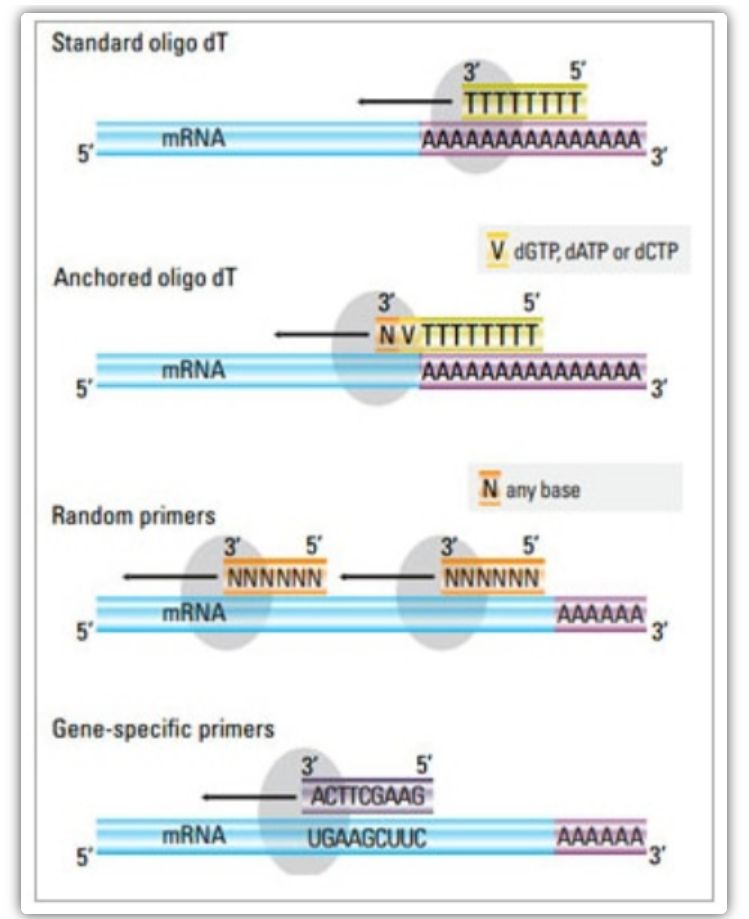
| ການຄັດເລືອກ primer | ໂຄງສ້າງແລະຫນ້າທີ່ | ຂໍ້ໄດ້ປຽບ | ຂໍ້ເສຍ |
| primer Oligo(dT) (ຫຼື oligo(dT) primer) | ຂະຫຍາຍການຫມຸນໃສ່ສານຕົກຄ້າງຂອງ thymine ຢູ່ຫາງ poly(A) ຂອງ mRNA;anchor oligo(dT) primer ມີ G, C, ຫຼື A ຢູ່ປາຍ 3′ (ສະຖານທີ່ສະມໍ) | ການສັງເຄາະ cDNA ເຕັມຄວາມຍາວຈາກ mRNA ຫາງ poly(A)
ນຳໃຊ້ໄດ້ເມື່ອມີວັດສະດຸເລີ່ມຕົ້ນໜ້ອຍລົງ
ສະຖານທີ່ຍຶດຫມັ້ນຮັບປະກັນວ່າ primer oligo(dT) ຜູກມັດກັບຫາງ 5′ poly(A) ຂອງ mRNA. | ເໝາະສຳລັບການຂະຫຍາຍພັນທຸກໍາທີ່ມີຫາງ poly(A) ເທົ່ານັ້ນ
ໄດ້ຮັບ cDNA ທີ່ຕັດອອກຈາກເວັບໄຊຕ່ໍາ *2 ໃນ poly(A)
ລໍາອຽງທີ່ຈະຜູກມັດກັບ 3′ ທ້າຍ*
*ຄວາມເປັນໄປໄດ້ນີ້ຈະຖືກຫຼຸດໜ້ອຍລົງຖ້າໃຊ້ primers oligo(dT) ທີ່ຖືກຍຶດໄວ້ |
| primer ແບບສຸ່ມ
| 6 ຫາ 9 ຖານໃນຄວາມຍາວ, ເຊິ່ງສາມາດຫມຸນໃສ່ຫຼາຍສະຖານທີ່ໃນລະຫວ່າງການຖອດຂໍ້ຄວາມ RNA | ເຊື່ອມຕໍ່ກັບ RNA ທັງຫມົດ (tRNA, rRNA, ແລະ mRNA)
ເຫມາະສໍາລັບການຖອດຂໍ້ຄວາມທີ່ມີໂຄງສ້າງຮອງທີ່ສໍາຄັນ, ຫຼືໃນເວລາທີ່ອຸປະກອນການເລີ່ມຕົ້ນຫນ້ອຍແມ່ນມີ
ຜົນຜະລິດ cDNA ສູງ | cDNA ຖືກຖອດຂໍ້ຄວາມຈາກ RNA ທັງໝົດ, ເຊິ່ງປົກກະຕິແລ້ວບໍ່ຕ້ອງການ ແລະອາດຈະເຈືອຈາງສັນຍານຂອງ mRNA ເປົ້າໝາຍ.
ໄດ້ຮັບການຕັດ cDNA |
| primers ລໍາດັບສະເພາະ | primers ແບບກຳນົດເອງທີ່ກຳນົດເປົ້າໝາຍສະເພາະຕໍ່ລຳດັບ mRNA | ຫ້ອງສະຫມຸດ cDNA ສະເພາະ
ປັບປຸງຄວາມອ່ອນໄຫວ
ການໃຊ້ primers reverse qPCR | ຈໍາກັດພຽງແຕ່ການສັງເຄາະຂອງ gene ເປົ້າຫມາຍດຽວ |
Reverse transcriptase
Reverse transcriptase ແມ່ນເອນໄຊທີ່ໃຊ້ RNA ເພື່ອສັງເຄາະ DNA.ການຖອດຂໍ້ຄວາມແບບປີ້ນກັບບາງອັນມີກິດຈະກໍາ RNase ແລະສາມາດທໍາລາຍສາຍພັນ RNA ໃນສາຍພັນປະສົມ RNA-DNA ຫຼັງຈາກການຖອດຂໍ້ຄວາມ.ຖ້າມັນບໍ່ມີກິດຈະກໍາ enzymatic RNase, RNaseH ສາມາດໄດ້ຮັບການເພີ່ມສໍາລັບປະສິດທິພາບ qPCR ສູງຂຶ້ນ.enzymes ທີ່ໃຊ້ທົ່ວໄປປະກອບມີ Moloney murine leukemia virus reverse transcriptase ແລະ avian myeloblastoma virus reverse transcriptase.ສໍາລັບ RT-qPCR, ມັນເຫມາະສົມທີ່ຈະເລືອກເອົາ reverse transcriptase ທີ່ມີ thermostability ສູງ, ດັ່ງນັ້ນການສັງເຄາະ cDNA ສາມາດປະຕິບັດຢູ່ໃນອຸນຫະພູມທີ່ສູງຂຶ້ນ, ຮັບປະກັນການຖ່າຍທອດ RNAs ສົບຜົນສໍາເລັດທີ່ມີໂຄງສ້າງມັດທະຍົມທີ່ສູງຂຶ້ນ, ໃນຂະນະທີ່ຮັກສາກິດຈະກໍາຢ່າງເຕັມທີ່ຂອງເຂົາເຈົ້າຕະຫຼອດຕິກິຣິຍາ, ເຮັດໃຫ້ຜົນຜະລິດ cDNA ສູງຂຶ້ນ.
ຜະລິດຕະພັນທີ່ກ່ຽວຂ້ອງ:
Foreasy M-MLV Reverse Transcriptase
ກິດຈະກໍາ RNase H ຂອງ reverse transcriptase
RNaseH ສາມາດທໍາລາຍສາຍ RNA ຈາກຄູ່ RNA-DNA, ຊ່ວຍໃຫ້ການສັງເຄາະຂອງ DNA ສອງສາຍຢ່າງມີປະສິດທິພາບ.ຢ່າງໃດກໍຕາມ, ເມື່ອໃຊ້ mRNA ຍາວເປັນແມ່ແບບ, RNA ອາດຈະຖືກທໍາລາຍກ່ອນໄວອັນຄວນ, ສົ່ງຜົນໃຫ້ cDNA ຖືກຕັດອອກ.ດັ່ງນັ້ນ, ມັນມັກຈະເປັນປະໂຫຍດທີ່ຈະຫຼຸດຜ່ອນກິດຈະກໍາ RNaseH ໃນລະຫວ່າງການ cloning cDNA ຖ້າຕ້ອງການການສັງເຄາະຂໍ້ຄວາມຍາວ.ໃນທາງກົງກັນຂ້າມ, ການຖອດຂໍ້ຄວາມແບບປີ້ນກັບກິດຈະກໍາ RNase H ມັກຈະເປັນປະໂຫຍດສໍາລັບຄໍາຮ້ອງສະຫມັກ qPCR ເພາະວ່າພວກມັນເສີມຂະຫຍາຍການລະລາຍຂອງ RNA-DNA duplexes ໃນໄລຍະຮອບວຽນທໍາອິດຂອງ PCR.
ການອອກແບບ primer
primers PCR ທີ່ໃຊ້ສໍາລັບຂັ້ນຕອນ qPCR ໃນ RT-qPCR ຄວນຈະຖືກອອກແບບໂດຍສະເພາະເພື່ອຂະຫຍາຍເສັ້ນທາງເຊື່ອມຕໍ່ exon-exon, ບ່ອນທີ່ primer ຂະຫຍາຍສາມາດຂະຫຍາຍຂອບເຂດຕົວຈິງຂອງ exon-intron.ເນື່ອງຈາກລໍາດັບ DNA genomic ທີ່ມີ intron ບໍ່ໄດ້ຖືກຂະຫຍາຍອອກ, ການອອກແບບນີ້ຫຼຸດລົງຄວາມສ່ຽງຂອງຜົນບວກທີ່ບໍ່ຖືກຕ້ອງທີ່ຖືກຂະຫຍາຍຈາກການປົນເປື້ອນ DNA ພັນທຸ ກຳ.
ຖ້າ primers ບໍ່ສາມາດຖືກອອກແບບເພື່ອແຍກຂອບເຂດ exons ຫຼື exon-exon, ມັນອາດຈະເປັນສິ່ງຈໍາເປັນເພື່ອປິ່ນປົວຕົວຢ່າງ RNA ດ້ວຍ RNase-free DNase I ຫຼື dsDNase ເພື່ອເອົາການປົນເປື້ອນຂອງ DNA genomic.
ການຄວບຄຸມ RT-qPCR
ການຄວບຄຸມທາງລົບຂອງການຖອດຂໍ້ຄວາມແບບປີ້ນກັບກັນ (-RT ການຄວບຄຸມ) ຄວນຖືກລວມເຂົ້າໃນການທົດລອງ RT-qPCR ທັງໝົດເພື່ອກວດຫາການປົນເປື້ອນຂອງ DNA (ເຊັ່ນ: ຜະລິດຕະພັນ DNA ພັນທຸ ກຳ ຫຼື PCR ຈາກປະຕິກິລິຍາທີ່ຜ່ານມາ).ການຄວບຄຸມນີ້ປະກອບດ້ວຍອົງປະກອບຕິກິຣິຍາທັງຫມົດຍົກເວັ້ນ reverse transcriptase.ເນື່ອງຈາກການຖອດຂໍ້ຄວາມແບບປີ້ນກັບກັນບໍ່ໄດ້ເກີດຂຶ້ນກັບການຄວບຄຸມນີ້, ຖ້າການຂະຫຍາຍ PCR ຖືກສັງເກດເຫັນ, ການປົນເປື້ອນຈາກ DNA ແມ່ນເປັນໄປໄດ້ຫຼາຍທີ່ສຸດ.
ເວລາປະກາດ: ສິງຫາ-02-2022








